Lexikon der Biologie: Onkogene
Onkogene [von *onko- , griech. gennan = erzeugen], Oncogene, Krebsgene, Tumorgene, onc-Gene, die zunächst bei Tumorviren, später aber auch im Kerngenom menschlicher und tierischer Zellen gefundenen Gene, deren Genprodukte unter bestimmten Bedingungen die Ursache für die Transformation gesunder Zellen zu Tumorzellen (Zelltransformation) sind (Krebs). Die viralen Onkogene (Abk. v-Onkogene) werden während des Infektionszyklus (Virusinfektion) onkogener Viren zusammen mit der übrigen Virus-DNA in das Genom der befallenen Zellen integriert. Bei den DNA-Tumorviren besitzen die Onkogene bzw. die von ihnen codierten Proteine nicht nur tumorinduzierende Funktion für die infizierten Zellen, sondern sind auch an der Replikation der viralen DNA beteiligt. Die Onkogene der Retroviren sind dagegen nur für die Transformation der infizierten Zellen verantwortlich und spielen bei der Virusreplikation keine Rolle. In einer Reihe von Tumorzellen, darunter auch in zahlreichen Tumorzellen des Menschen, wurden aktiv transformierende zelluläre Onkogene (Abk. c-Onkogene) gefunden, die sequenzhomolog (Sequenzhomologie) mit bereits charakterisierten Onkogenen aus Retroviren sind. Die aktiv transformierenden viralen (in Retroviren) und zellulären Onkogene leiten sich von sog. Protoonkogenen ab, die stets als normaler, nicht pathogener Bestandteil im Genom gesunder Zellen vorliegen. Die Umwandlung von Protoonkogenen zu aktiv transformierenden zellulären Onkogenen erfolgt durch verschiedenartige somatische Mutationen, induziert z.B. durch Umweltkarzinogene (cancerogen). Das in einer gesunden Zelle in nur geringem Umfang transkribierte Protoonkogen kann z.B. durch Translokation innerhalb des Genoms (gefunden für das sog. c-myc-Onkogen; c-myc) oder durch Insertion von Retrovirus-DNA, die selbst kein Onkogen trägt, unter die Kontrolle eines starken Promotors gelangen, so daß nun mehr Genprodukt gebildet wird. Auch Mutationen innerhalb des normalerweise für das Protoonkogen zuständigen Promotors können zur Änderung der Genproduktmenge führen. Ebenso kann die Amplifikation (Genamplifikation) eines Protoonkogens zu einer übermäßigen Expression des Gens (Genexpression) und somit zur Tumorinduktion führen. Eine weitere Möglichkeit wurde beim sog. c-ras-Onkogen aus dem menschlichen EJ-Blasenkarzinom gefunden: das aktiv transformierende Onkogen unterscheidet sich von dem in gesunden Zellen vorliegenden Protoonkogen durch einen einzigen Basenaustausch innerhalb des codierenden Bereichs (eine Punktmutation; vgl. Abb. 1 ). – Die Funktion der Onkogenprodukte im Zellstoffwechsel und die Wirkung ihrer veränderten Expression nach dem Übergang vom Protoonkogen zum aktiven transformierenden viralen oder zellulären Onkogen sind in vielen Fällen noch ungeklärt. Es erscheint jedoch sicher, daß die Protoonkogene in der Regel entscheidend an der Regulation von Zellwachstums- und Zelldifferenzierungsprozessen (Differenzierung) beteiligt sind, so daß Mutationen dieser Gene eine Störung im Signaltransduktionsweg hervorrufen, die zur Tumorentstehung führen kann. – Die Onkogene können nach ihrer normalen zellulären Funktion in 5 Gruppen eingeteilt werden: 1) Wachstumsfaktoren ( vgl. Tab. 1 ), 2) Rezeptoren ( vgl. Tab. 2 ), 3) Proteine des Signaltransduktionswegs im Cytoplasma ( vgl. Tab. 3 ), 4) Transkriptionsfaktoren ( vgl. Tab. 4 ), 5) Tumorsuppressorgene ( vgl. Tab. 5 ). Onkogene, die zu den Wachstumsfaktoren gehören, beeinflussen die Zelltransformation u.a. dadurch, daß sie überexprimiert (Überexpression) werden und somit den second-messenger-Mechanismus permanent aktivieren. Z.B. konnte für einige virale Onkogene aufgrund ausgeprägter Sequenzverwandtschaften gezeigt werden, daß sie Abkömmlinge von zellulären Wachstumsfaktoren bzw. von deren Rezeptoren sind (z.B. v-sis/platelet derived growth factor [PDGF]; v-erbB/epidermal growth factor-(EGF-)Rezeptor [epidermal growth factor]). Die permanente Aktivierung von second-messenger Mechanismen trifft ebenfalls auf einige Onkogene der Klasse der Rezeptoren zu. Darüber hinaus können veränderten Rezeptoren die extrazellulären Liganden-Bindungs-Domänen fehlen, so daß sie ohne die Bindung eines Wachstumsfaktors permanent ein Signal an die Proteine des Signaltransduktionswegs weiterleiten. Veränderte Proteine des Signaltransduktionswegs (Signaltransduktion; vgl. Abb. 2 ) tragen u.a. zur Zelltransformation bei, indem sie in einem aktivierten, nicht mehr regulierbaren Zustand vorliegen, so daß z.B. durch Phosphorylierungsreaktionen (Phosphorylierung) ununterbrochen weitere zelluläre Proteine aktiviert werden. In einigen Fällen handelt es sich bei derartigen von Onkogenen codierten Proteinen um Tyrosin-spezifische Proteinkinasen (Tyrosin-Kinase). Die onkogen wirkenden Transkriptionsfaktoren greifen störend in die Regulation der Genexpression ein. Sie weisen häufig Deletionen auf, die eine Kooperation mit anderen Transkriptionsfaktoren und zellulären Proteinen verhindern. Die Tumorsuppressorgene, die normalerweise das Zellwachstum inhibieren, scheinen eine sehr wichtige, bisher unterschätzte Rolle bei der Krebsentstehung (Karzinogenese) zu spielen. Sie übernehmen bei der Tumorentstehung eine andere Rolle als die restlichen Onkogene der Gruppen 1–4. Während die zuletzt genannten Onkogene tumorinduzierend wirken, indem sie überexprimiert werden und/oder der zellulären Regulation entzogen sind, tragen mutierte Tumorsuppressorgene zur Zelltransformation dann bei, wenn ihre Genprodukte funktionslos sind oder nicht mehr gebildet werden. Im Gegensatz zu den übrigen Onkogenen, die eine dominante Wirkungsweise ausüben, weisen die meisten Tumorsuppressorgene eine rezessive Funktionsweise auf, d.h., die Mutation eines Allels beeinflußt noch nicht die Suppressoreigenschaft. Dafür ist die Mutation beider Allele notwendig. – Zu den 5 Gruppen der Onkogene zählen sowohl die zellulären Onkogene (c-onc) als auch die viralen Onkogene (v-onc) der Retroviren ( vgl. Tab. 6 ), die sich von zellulären Protoonkogenen ableiten und Bestandteil des retroviralen Genoms sind ( vgl. Abb. 3 ). Man nimmt an, daß die Retroviren diese zellulären Gene über Infektionszyklen in Wirtszellen erworben haben. Eine weitere Gruppe sind die Onkogene der DNA-Viren ( vgl. Tab. 6 ), die nicht homolog zu zellulären Genen sind und u.a. eine Funktion bei der Virusvermehrung übernehmen. Ihre tumorinduzierende Wirkung beruht auf einer zum Teil noch ungeklärten Interaktion mit zellulären Kontrollmechanismen, wodurch die normale Regulation der Zellteilung und -differenzierung gestört wird. – Durch die Untersuchung an Onkogenen wurden Einblicke in einen, möglicherweise aber den auslösenden Teilschritt der Tumorentstehung gewonnen, nämlich in die Veränderung der betroffenen Zellen aufgrund somatischer Mutation von Zellzyklus-regulierenden Genen. Nach wie vor geht man allerdings davon aus, daß zur Bildung eines Tumors mehrere Schritte nötig sind, daß also zur Entstehung eines aktiv transformierenden Onkogens weitere Änderungen (z.B. Induktion durch sog. Tumorpromotoren) hinzukommen müssen. Eine vollständige Transformation von Zellen in vitro (z.B. von embryonalen Ratten-Fibroblasten) erfordert meist eine Kooperation verschiedener Onkogene. Eine Gruppe von Onkogenen (dazu gehören: v-myc und c-myc, Adenovirus E1A, Polyoma large-T) führt zur Immortalisierung der Zellen, die dann durch die Einwirkung einer zweiten Gruppe von Onkogenen (z.B. virale und aktivierte zelluläre ras-Gene [Ras-Proteine], Polyoma middle-T) vollständig transformiert werden können. – Zu menschlichen Protoonkogenen homologe DNA-Sequenzen wurden nicht nur in Wirbeltieren, sondern auch im Genom von Drosophila, von Hefen und Pflanzen identifiziert. Onkogene sind offensichtlich im Organismenreich weit verbreitet und strukturell stark konserviert, was ein weiterer Hinweis auf ihre zentrale Bedeutung für die Regulation des Wachstums gesunder Zellen ist. Adenoviren, Antionkogene, Apoptose, Bishop (M.J.), BRCA1, c-mos, Gentechnologie, p53-Protein, Varmus (H.E.).
G.St./E.G./S.Kl.
Onkogene
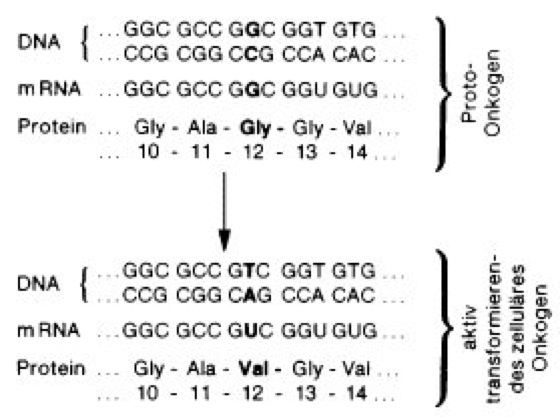
Abb. 1: Das aktiv transformierende zelluläre c-ras Onkogen in menschlichen EJ-Blasenkarzinomzellen entsteht aus seinem Protoonkogen durch einen einzigen Basenaustausch: anstelle eines Guanins (G) an der 2. Position des 12. Aminosäuren-Codons steht ein Thymin (T), so daß im zugehörigen Protein statt der Aminosäure Glycin die Aminosäure Valin vorliegt.
Onkogene
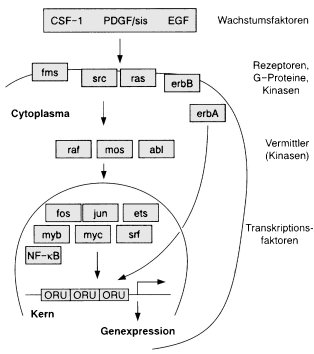
Abb. 2: Die meisten Onkogene bzw. die ihnen zugrundeliegenden Protoonkogene sind an Signaltransduktions-Mechanismen aus der Umgebung in das Innere von Zellen beteiligt, wobei im allgemeinen letztlich die Genexpression bestimmter Gene oder Gengruppen über regulatorisch wirksame DNA-Abschnitte (ORU = oncogene responsive units) beeinflußt wird. Aus jeder der Stufen der Signaltransduktion sind entsprechende Onkogene bekannt. Einige Beispiele mit den jeweils allgemein benutzten Bezeichnungen sind in ihrer relativen Lokalisation in einer Modell-Zelle dargestellt.
Onkogene
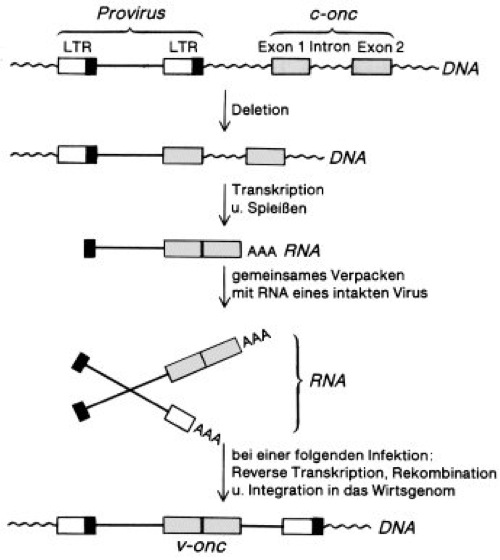
Abb. 3: Aufnahme von Protoonkogenen in das Genom von Retroviren:
Zunächst integriert die DNA eines Retrovirus in der Nähe eines Protoonkogens in das Genom der infizierten Zelle. Durch Deletion rückt daraufhin die Provirus-DNA in die Nähe des Protoonkogens, wobei mehr oder weniger große 3'-terminale Bereiche des Provirus und 5'-terminale Bereiche des Protoonkogens verlorengehen. Das gemeinsame Transkriptionsprodukt des 5'-Anteils des Provirus und des Protoonkogens können schließlich zusammen mit intakter Virus-RNA in ein Virus-Partikel verpackt werden (jedes Retrovirus-Partikel enthält zwei RNA-Moleküle). Rekombination auf DNA-Ebene (bei einer späteren Infektion) kann dazu führen, daß das Protoonkogen in ein bislang intaktes Virus-Genom aufgenommen wird. Da bei dieser Rekombination wesentliche Teile des Virus-Genoms verlorengehen, kann die Vermehrung dieses Virus nur noch mit Hilfe anderer, intakter Retroviren erfolgen, die noch sämtliche für die Replikation und Verpackung der Virus-RNA notwendigen Funktionen codieren (Helfer-Viren). Wie die in das Virus-Genom integrierten Protoonkogene zu aktiv transformierenden viralen Onkogenen werden, ist nicht endgültig geklärt. Eine Möglichkeit ist, daß das Protoonkogen unter der Kontrolle des in der LTR-Region des Retrovirus befindlichen Promotors erheblich stärker transkribiert wird. Wellenlinien symbolisieren Wirts-DNA, AAA steht für das poly(A)-Ende der mRNA; LTR = Abk. für Long Terminal Repeat (LTR; Retroviren).
Onkogene
Tab. 1: Zelluläre Onkogene; 1. Klasse: Wachstumsfaktoren
| ||||||
| durch Überexpression permanente Aktivierung der Rezeptoren | ||||||
| cripto | verwandt mit EGF, TGFα | |||||
| EGF (epidermal growth factor) | Wachstumsfaktor der EGF-Familie, verwandt mit cripto, TGFα | verschiedene Tumore | ||||
| b-FGF (basischer fibroblast growth factor) | Wachstumsfaktor der FGF-Familie | wird von menschlichen Melanomazellen exprimiert, nicht aber von Melanocyten | ||||
| FGF-3 | int-2 | site of frequent virus integration | Wachstumsfaktor der FGF-Familie | a) Überexpression durch Insertion proviraler DNA b) Genamplifikation wird angenommen | a) Brusttumore bei Mäusen b) u.a. Brusttumore beim Menschen | |
| FGF-4 | K-FGF/HST | Fundort: Kaposi-Sarkom | Wachstumsfaktor der FGF-Familie | verschiedene Tumore, u.a. Brust- und Magentumore | ||
| FGF-5 | Wachstumsfaktor der FGF-Familie | verschiedene Tumore | ||||
| TGFα (transforming growth factor) | bindet an den EGF-Rezeptor | |||||
| int-1 | site of frequent virus integration | Wachstumsfaktor? | Überexpression durch Insertion proviraler DNA | Brusttumore bei Mäusen |
Onkogene
Tab. 2: Zelluläre Onkogene; 2. Klasse: Rezeptoren (-R)
| ||||||
| durch Überexpression oder fehlende Liganden-Bindungsdomäne permanente Aktivierung des second-messenger-Mechanismus | ||||||
| bek | FGF-R2 | Mitglied der FGF-R-Familie, Protein-Tyrosin-Kinase-Rezeptor | Genamplifikation oder Überexpression | Magentumore | ||
| erbB-1 (EGF-R, epidermal growth factor receptor, Protein-Tyrosin- Kinase-Rezeptor) | EGF-R | Erythroblastosis | Mitglied der EGF-R-Familie, bindet EGF, TGFα, Amphiregulin (AR) | Genamplifikation oder Überexpression | verschiedene Tumore, u.a. Brust-, Gehirn- und Schilddrüsentumore | |
| erbB-2 (EGF-R, epidermal growth factor receptor, Protein-Tyrosin-Kinase-Rezeptor) | a) neu b) HER-2 | Erythroblastosis | a) Mitglied der EGF-R-Familie b) menschlicher Rezeptor der EGF-Familie, bindet spezifisch Heregulin | Genamplifikation oder Überexpression | verschiedene Tumore, u.a. Brust-, Magen-, Pankreas- und Ovarialtumore | |
| erbB-3 (EGF-R, epidermal growth factor receptor, Protein-Tyrosin- Kinase-Rezeptor) | Erythroblastosis | Mitglied der EGF-R-Familie | Überexpression | Brusttumore | ||
| 5-HTlc | Mitglied der Serpentin-Familie, Serotonin-Rezeptor | |||||
| kit | SLF | Protein-Tyrosin-Kinase-Rezeptor, homolog zu PDGF-R (platelet derived growth factor-Rezeptor) und CSF1-R (colony stimulating factor 1-Rezeptor), bindet SCF (stem cell factor) | vermutlich bei AML (acute myeloid leukemia) | |||
| mas | Mitglied der Serpentin-Rezeptor- Familie, Angiotensin-Rezeptor, vermutlich Rezeptor für Neurotransmitter ohne Kinasefunktion | |||||
| met | Protein-Tyrosin-Kinase-Rezeptor, bindet HGF (hepatocyte growth factor) | Genamplifikation oder Überexpression | Magentumore | |||
| ptc-1 | Papillary Thyroid Carcinoma | Fusionsprotein aus der 3'-Protein- Tyrosin-Kinase-Domäne von ret und einem weiteren Gen | Rearrangement | Schilddrüsentumore | ||
| ptc-2 | Papillary Thyroid Carcinoma | Fusionsprotein aus der 3'-Protein-Tyrosin-Kinase-Domäne von ret und einem weiteren Gen, das nicht dem von ptc-1 entspricht | Rearrangement | Schilddrüsentumore | ||
| ret | Protein-Tyrosin-Kinase-Rezeptor | Rearrangement | Schilddrüsentumore | |||
| sea | Protein-Tyrosin-Kinase-Rezeptor, Insulin-Rezeptor | |||||
| trk | NGF-R | Protein-Tyrosin-Kinase-Rezeptor, NGF (nerve growth factor)-Rezeptor | Rearrangement | Schilddrüsen- und Dickdarmtumore | ||
| trk-T1 und -T2 | Protein-Tyrosin-Kinase-Rezeptor | Rearrangement | Schilddrüsentumore |
Onkogene
Tab. 3: Zelluläre Onkogene; 3. Klasse: Proteine des Signaltransduktionswegs im Cytoplasma
| ||||||
| durch Mutationen befinden sich die Enzyme in einem aktivierten Zustand, Enzymaktivität ist nicht mehr regulierbar | ||||||
| abl | Abelson Murine Leukemia Virus | Protein-Tyrosin-Kinase, Erhöhung der Kinaseaktivität durch Fusion mit dem bcl-Gen | Rearrangement (Translokation) | verschiedene Leukämieformen | ||
| Cyclin D1 | PRAD1 | Parathyroid Adenomatosis 1 | Zellzyklusprotein | Rearrangement | Tumore der Nebenschilddrüse, B-lymphoide Tumore, vermutlich auch Brusttumore | |
| dbl | Guanin-Releasing- Faktor, Mitglied der rho-Familie | |||||
| ect2 | interagiert mit rho und ras in Funktion eines Guanin-Releasing- Faktors | Deletionen | ||||
| fps | Protein-Tyrosin-Kinase | |||||
| lck | Protein-Tyrosin-Kinase, Mitglied der src-Familie | Punktmutationen | Tumore der Thymusdrüsen | |||
| pim-1 | Protein-Serin-Kinase | |||||
| raf | Serin-Threonin-Kinase | Deletionen oder Punktmutationen | Lungentumore | |||
| rap1A | a) Krev-1 b) smg p21A | verwandt mit ras-Familie | vermutlich Down-Regulation | verschiedene Sarkome | ||
| rap1B | smg p21B | verwandt mit ras-Familie | verschiedene Sarkome | |||
| ras | GTPase (GTP-bindendes Protein) | Punktmutationen | Lungen-, Dickdarm- und Pankreastumore | |||
| H-ras | Ha-ras | Harvey Murine Sarcoma Virus | GTPase (GTP-bindendes Protein) | Punktmutationen | Lungen-, Dickdarm-, Haut- und Pankreastumore | |
| T24-H-ras | GTPase (GTP-bindendes Protein) | Blasentumore |
| ||||||
| N-ras | Neuroblastoma | GTPase (GTP-bindendes Protein) | Amplifikation oder Punktmutationen | Neuroblastome, Haut- und Schilddrüsentumore |
Onkogene
Tab. 4: Zelluläre Onkogene; 4. Klasse: Transkriptionsfaktoren
| ||||||
| durch Mutationen ist die Interaktion mit anderen Proteinen und/oder der DNA verändert, gestörte Regulation der Genexpression | ||||||
| fli-1 | Mitglied der ets-Familie, Transkriptionsfaktor der PEA-3-Bindungsstelle | Überexpression durch Insertion proviraler DNA | Erythroleukämie bei Mäusen | |||
| fos | bindet als Heterodimer zusammen mit c-jun an die AP-1-Bindungsstelle, besitzt Leucin-Zipper-Motiv | Deletionen oder Überexpression | Osteosarkom bei transgenen Mäusen | |||
| IRF-2 (interferon regulation factor) | Antagonist zu IRF-1, einem Transkriptionsaktivator des Typ I-Interferons | |||||
| jun | bindet sowohl als Homo- als auch als Heterodimer zusammen mit c-fos an die AP-1-Bindungsstelle, besitzt Leucin-Zipper-Motiv | Deletionen oder Überexpression | ||||
| myc | Myelomatosis | sequenzspezifischer Transkriptionsfaktor, besitzt Helix-Loop-Helix-Motiv | Rearrangement, Amplifikation oder Überexpression | Burkitt-Lymphom und B- und T-Zell-Lymphome, Brust- und Lungentumore | ||
| L-myc | Myelomatombs | sequenzspezifischer Transkriptionsfaktor, besitzt Helix-Loop-Helix-Motiv | Amplifikation | Lungentumore | ||
| N-myc | Myelomatosis | sequenzspezifischer Transkriptionsfaktor, besitzt Helix-Loop-Helix-Motiv | Amplifikation | Lungentumore und Neuroblastome | ||
| Pu.1 | SPI-1 | Mitglied der ets-Familie |
Onkogene
Tab. 5: Zelluläre Onkogene; 5. Klasse: Tumorsuppressorgene
| ||||||
| durch Mutation sind Genprodukte funktionslos, fehlende Inhibition des Zellwachstums | ||||||
| APC | Adenomatosis Polyposis coli | assoziiert mit den beiden zellulären Proteinen α- und β-Catenin | Deletionen | Dickdarmtumore | ||
| BRCA1 | Brust- und Ovarialtumore | |||||
| DCC | deleted in colon carcinoma | zeigt Strukturähnlichkeiten zu neuralen Adhäsionsmolekülen | Deletionen | Dickdarm- und Gehirntumore | ||
| NF1 | Neurofibromatose | GTPase-aktivierendes Protein, besitzt Homologien zur ras-GTPase | Deletionen | Neurofibrosarkome | ||
| nm 23H-1 | Nucleotid-Diphosphat- Kinase | |||||
| p53 | Phosphoprotein, das an DNA bindet, vermutlich negativer Regulator des Zellwachstums und der Zellteilung | Deletionen und Punktmutationen | Dickdarm-, Lungen-, Knochen- und Brusttumore, Li-Fraumeni-Syndrom | |||
| PKR | Proteinkinase, die durch Doppelstrang-RNA aktiviert wird | |||||
| Rb | Retinoblastom | Phosphoprotein, das an DNA bindet, vermutlich negativer Regulator des Zellwachstums, der Zellteilung und der c-fos-Expression | Deletionen | Retinoblastome, Knochen-, Brust-, Blasen- und Lungentumore | ||
| VHL | Von-Hippel-Lindau-Syndrom | vermutlich Zelloberflächenprotein mit Funktion bei der Zelladhäsion und Signaltransduktion | Rearrangement | Angioblastome des Gehirns | ||
| WT1 | Wilms-Tumor | Transkriptionsfaktor, besitzt Zink-Finger-Motiv | Wilms-Tumor (Nierentumor) |
Onkogene
Tab. 6: VIRALE ONKOGENE
| ||||
| 1) Retrovirale Onkogene | ||||
| 1. Klasse: Wachstumsfaktoren | ||||
| v-sis | Simian Sarcoma Virus | β-Kette von PDGF (platelet derived growth factor) | – | |
| 2. Klasse: Rezeptoren | ||||
| v-env-sea | Avian Erythroblastosis Virus (AEV S13) | Fusionsprotein aus dem viralen Hüllprotein env und dem zellulären Protein sea | – | |
| v-erbB | Avian Erythroblastosis Virus (AEV ES4) | verkürzte Form des Hühner-EGF-Rezeptors, Protein-Tyrosin-Kinase-Rezeptor | – | |
| v-fms | Feline Sarcoma Virus (SM-FeSV) | Mutante des CSF1-Rezeptors (colony stimulating factor 1), Protein-Tyrosin-Kinase-Rezeptor | – | |
| v-kit | Acute Feline Retrovirus (HZ4-FeSV) | Protein-Tyrosin-Kinase-Rezeptor | + | |
| v-ros | Protein-Tyrosin-Kinase-Rezeptor | – | ||
| 3. Klasse: Proteine des Signaltransduktionswegs im Cytoplasma | ||||
| v-abl | Abelson Murine Leucemia Virus | Protein-Tyrosin-Kinase | + | |
| v-akt | Acute Transforming Retrovirus AKT 8 | Serin-Threonin-Kinase, Fusionsprotein aus gag und dem zellulären akt-Gen | – | |
| v-crk | assoziiert mit Protein-Tyrosin-Kinase-Aktivität, bindet über SH-2/3-Domänen Proteine mit Phosphotyrosinresten | – | ||
| v-fgr | Protein-Tyrosin-Kinase, Mitglied der src-Familie | – | ||
| v-fps | Fujinama Sarcoma Virus | Protein-Tyrosin-Kinase | + | |
| v-mos | Moloney Sarcoma Virus | Serin-Threonin-Kinase | – | |
| v-raf | Murine Sarcoma Virus | Serin-Threonin-Kinase | + | |
| v-H-ras | Harvey Murine Sarcoma Virus | GTPase | + | |
| v-K-ras | Kirsten Murine Sarcoma Virus | GTPase | + | |
| v-R-ras | Rasheed Murine Sarcoma Virus | GTPase | – | |
| v-src | Rous Sarcoma Virus | Protein-Tyrosin-Kinase, Mitglied der src-Familie | – | |
| v-yes | Protein-Tyrosin-Kinase | – | ||
| 4. Klasse: Transkriptionsfaktoren | ||||
| v-cbl | Murine Cas NS-1 Retrovirus | ? | – | |
| v-erbA | Avian Erythroblastosis Virus | vermutlich Transkriptionsrepressor | – | |
| v-ets | Avian Retrovirus E26 | Fusionsprotein aus gag, v-myb und v-ets | – | |
| v-fos | Murine Osteosarcoma-Viren: FBJ MuSV und FBR MuSV | bindet an die AP-1-Bindungsstelle | + | |
| v-jun | Avian Sarcoma Virus 17 (ASV-17) | + | ||
| v-maf | Avian Retrovirus AS 42 | – | ||
| v-myb | Avian Myeloblastosis Virus (AMV) | sequenzspezifischer Transkriptionsfaktor | – | |
| v-myc | Avian Myelocytomatosis Virus (MC 29) | + | ||
| v-qin | Avian Sarcoma Virus 31 | – | ||
| v-rel | Avian Reticuloendotheliosis Virus (REV-T) | verwandt mit Transkriptionsfaktor NF-κB | – | |
| 2) Onkogene aus DNA-Viren | ||||
| E1A | Adenovirus | reguliert die Transkription, bindet das Genprodukt des Tumorsuppressorgens Rb | – | |
| E1B | Adenovirus | bindet das Genprodukt des Tumorsuppressorgens p53 | – | |
| E6 | Papillomavirus | bindet das Genprodukt des Tumorsuppressorgens p53 | – | |
| E7 | Papillomavirus | bindet das Genprodukt des Tumorsuppressorgens Rb | – | |
| SV40 large T-Antigen | SV40-Virus | initiiert die DNA-Synthese, reguliert die Transkription, bindet die Genprodukte der Tumorsuppressorgene p53 und Rb | – | |
| PV large T-Antigen | Polyomavirus | initiiert die DNA-Synthese, reguliert die Transkription, bindet das Genprodukt des Tumorsuppressorgens p53 | – | |
| PV middle T-Antigen | Polyomavirus | bindet und stimuliert die Protein-Tyrosin-Kinasen src und yes sowie PI3-K (Phosphatidylinositol-3-Kinase) und die Phosphatase PP2A | – | |
| PV small T-Antigen | Polyomavirus | ? | – |
Schreiben Sie uns!