Lexikon der Biologie: Proteine
Proteine [von griech. prōtos = der erste (Stoff)], Eiweiße, Eiweißstoffe, Eiweißkörper, die vorwiegend aus den 20 proteinogenen α-L-Aminosäuren (Farbtafel) durch Peptidbindungen (Peptide) primär linear aufgebauten Polymere (Biopolymere), die jedoch aufgrund kovalenter, ionischer, polarer und hydrophober Wechselwirkungen (chemische Bindung) in der Lage sind, hochspezifische, den jeweiligen Funktionen angepaßte räumliche Strukturen auszubilden. Die Bezeichnung „Eiweiß“ ist historisch bedingt und geht auf die ursprüngliche Isolierung aus dem Hühnereiweiß (Hühnerei) zurück. Schon relativ früh zeigte sich jedoch, daß Eiweiße Bestandteile praktisch aller lebenden Zellen sind und daß sie aufgrund ihrer Strukturvielfalt an nahezu allen Lebensprozessen wesentlichen Anteil haben (Leben). Deshalb wird heute allgemein die von Berzelius 1838 geprägte Bezeichnung „Protein“ (von griech. prōteios = erstrangig) der älteren Bezeichnung „Eiweiß“ vorgezogen. Während z.B. eine Zelle von Escherichia coli ca. 3000 verschiedene Proteine enthält, finden sich im menschlichen Organismus mehr als 100.000 unterschiedliche Proteine.
Aufbau und Struktur: Der Aufbau der Proteine aus den 20 verschiedenen Aminosäuren ist schriftartig, d.h. mit einer für jedes Protein charakteristischen Reihenfolge der einzelnen Aminosäuren, die auch als Reste bezeichnet werden (Primärstruktur, Aminosäuresequenz; s.u.). Auch die Kettenlänge ist für jedes einzelne Protein charakteristisch und bewegt sich zwischen nur wenigen Aminosäureresten (z.B. 21 in der A-Kette des Insulins; Hormone ) und über 1000 Aminosäureresten, was relativen Molekülmassen von 2000 bis über 100.000 (makromolekulare chemische Verbindungen) entspricht. Alle Peptidbindungen eines Proteins enthalten denselben Richtungssinn, indem sie sich durch „Kopf-an-Schwanz-Verknüpfung“ der einzelnen Aminosäuren ableiten. Daraus resultiert letztlich auch die durch den Aminoterminus und den Carboxylterminus definierte Polarität jeder Proteinkette (vereinbarungsgemäß schreibt man den Aminoterminus immer an den Anfang, den Carboxylterminus an das Ende einer Aminosäuresequenz). Aufgrund der Verknüpfung der einzelnen Aminosäuren durch Peptidbindungen besteht eine Proteinkette aus regelmäßig sich wiederholenden Einheiten, dem sog. Rückgrat, und den verschiedenen Seitenketten der einzelnen Aminosäuren. Die reinen Proteine enthalten entsprechend ihrem Aufbau aus den Aminosäuren die chemischen Elemente (Bioelemente) C (Kohlenstoff), H (Wasserstoff), O (Sauerstoff), N (Stickstoff) (in jedem Aminosäurerest enthalten) und S (Schwefel; nur in Cystein und Methionin enthalten). Neben diesen 20 proteinogenen Aminosäuren enthalten bestimmte Proteine in meist geringen Anteilen auch modifizierte Aminosäuren (z.B. N-Formyl-Methionin am Aminoterminus bakterieller Proteine, Hydroxyprolin und Hydroxylysin in Kollagen), Metalle (Metalloproteine), prosthetische Gruppen, Phosphatreste (Phosphoproteine) sowie in unterschiedlich starken Anteilen Lipide (Lipoproteine), Nucleinsäuren (Nucleoproteine; Viren), Zuckerreste (Glykoproteine) und Polysaccharide (Proteoglykane). Solche in der Regel co- oder posttranslational eingeführten Modifikationen (posttranslationale Proteinmodifikation) haben wesentlichen Einfluß auf die Eigenschaften von Proteinen. Sie beinflussen z.B. deren Stabilität, Löslichkeit, Hydrophobizität und – im Falle reversibler Modifikationen – deren Aktivität. Die Aktivität vieler Proteine, z.B. von Verdauungsenzymen (Verdauung) oder Faktoren der Blutgerinnung, kann außerdem durch Spaltung inaktiver Vorstufen modifiziert werden. Meist nur locker gebundene und daher austauschbare niedermolekulare Komponenten von Proteinen sind die zur Neutralisation geladener Gruppen erforderlichen Ionen sowie bei Enzymproteinen (Enzyme) die Coenzyme, Inhibitoren und Aktivatoren.
Als Primärstruktur der Proteine ( vgl. Abb. 1 ) – identisch mit dem Begriff Aminosäuresequenz – bezeichnet man die lineare, schriftartige Reihenfolge der einzelnen Aminosäurereste jedes Proteins. Sie ist genetisch festgelegt durch die Basensequenz der jeweiligen Gene (Desoxyribonucleinsäuren, Ein-Gen-ein-Enzym-Hypothese, genetischer Code, Ribonucleinsäuren, Translation). Die enorme Vielfalt möglicher Primärstrukturen läßt sich durch folgende Überlegung veranschaulichen: Nach den Gesetzen der Kombinatorik ergeben sich für die aus n Aminosäuren aufgebauten Ketten 20n theoretische Möglichkeiten. Das bedeutet, daß allein für die relativ geringe Kettenlänge von 100 Aminosäuren 20100 = 10130 theoretische Möglichkeiten existieren, wovon in den heute lebenden Organismen (aber auch, wenn man alle im Laufe der Erdgeschichte aufgetretenen Spezies einbezieht) nur ein verschwindend kleiner Bruchteil realisiert wurde (in Anbetracht der für das gesamte Universum auf etwa 1080 geschätzten Zahl von Elementarteilchen ist die Zahl 10130 „überastronomisch hoch“). Die Ähnlichkeit der Primärstrukturen homologer Proteine (Sequenzhomologie), insbesondere solcher, die in höherem Maße konserviert sind, wie z.B. Histone oder Cytochrome, hat sich als Kriterium zur Aufstellung von Sequenzstammbäumen – analog zu den Stammbäumen auf der Basis phänotypischer Merkmale (Stammbaum) – bewährt und gibt Einblick in die molekulare Evolution. Da die Primärstruktur wesentlich die Faltung und damit die übergeordneten Strukturen von Proteinen (s.u.) beeinflußt, bietet deren Aufklärung die Möglichkeit zur Strukturvorhersage und gibt Einblicke in die Wirkungsweise von Proteinen. Außerdem sind in der Aminosäuresequenz Signale für die Lokalisierung von Proteinen in der Zelle (Proteintransport) und für die Prozessierung (z.B. Glykosylierung, proteolytische Aktivierung) enthalten. Veränderungen der Primärstruktur von Proteinen können zu Krankheiten führen, gezielte Veränderungen der Primärstruktur dienen der Erzeugung von Proteinen mit entsprechend veränderten Eigenschaften und Wirkungen (protein engineering).
Als Sekundärstrukturen der Proteine faßt man alle durch Wasserstoffbrückenbindungen (Wasserstoffbrücke) bedingten, periodisch wiederkehrenden Überstrukturen zusammen, wobei sich durch H-Brücken zwischen Sequenz-nahen Resten Schrauben- oder Helixstrukturen (Helices), durch H-Brücken zwischen Sequenz-fernen parallel oder antiparallel liegenden Peptidketten die Faltblattstrukturen ausbilden. Letztere können auch zwischen verschiedenen Peptidketten entstehen. Zu den Sekundärstrukturen ist schließlich auch die aus 3 parallelen Ketten aufgebaute Schraubenstruktur des Kollagens zu rechnen, da auch hier regelmäßig wiederkehrende Wasserstoffbrückenbindungen die Stabilität bewirken. Die am häufigsten vorkommende Schraubenstruktur ist die α-Helix (Alpha-Helix; vgl. Abb. 1 und vgl. Abb. 2 ), die unabhängig vom Windungssinn, der jedoch in natürlichen Polypeptiden rechtsdrehend ist, auch als α-Struktur bezeichnet wird. Die unterschiedlichen Substituenten der einzelnen Aminosäurereste ragen in den Schraubenstrukturen nach außen und können daher mit dem umgebenden Medium bzw. mit den Substituenten entfernterer Aminosäuren derselben Kette oder auch Substituenten anderer Ketten in Wechselwirkung treten. Die den schriftartigen Charakter bedingenden Substituenten (Seitenreste) sind in den helikalen Proteinstrukturen also von außen zugänglich – im Gegensatz zur DNA-Doppelhelix, in der die Basen, die hier den schriftartigen Charakter bedingen, im Innern „vergraben“ sind ( ä Desoxyribonucleinsäuren III ). Die Aminosäure Prolin besitzt als einzige der 20 Aminosäuren eine in einen Ring eingebundene und daher sekundäre Aminogruppe ( ä Aminosäuren ) und kann daher nicht die für helikale H-Brücken erforderliche Konfiguration einnehmen. Aus diesem Grund werden α-Strukturen an allen Prolin-Positionen einer Peptidkette unterbrochen (Funktion von Prolin als sog. Helix-Unterbrecher), was für das Abknicken von Helixstrukturen bei der Ausbildung von Tertiärstrukturen (s.u.) von Bedeutung ist. Durch inter- oder intramolekulare antiparallele Aneinanderlagerung von Peptidketten entstehen Faltblattstrukturen, die man als β-Strukturen (Beta-Faltblatt) bezeichnet (Antiparallelität). Die Substituenten der einzelnen Aminosäurereste ragen bei den β-Strukturen alternierend in entgegengesetzte Richtung bezüglich der Faltblattebene ( vgl. Abb. 3 ). Die für die Ausbildung antiparalleler Faltblattstränge notwendige abrupte Umkehr der Richtung einer Polypeptidkette kommt an sog. β-Schleifen zustande, die in ihrer Struktur den Haarnadelschleifen von Nucleinsäuren ähnlich sind. Sekundärstrukturen bilden sich in der Regel nicht über die gesamte Länge von Peptidketten aus. So ist z.B. Myoglobin zu über 75% aus α-Strukturen aufgebaut, was einem relativ hohen Sekundärstrukturgehalt entspricht, während Chymotrypsin einen α-Helixgehalt von nur 8% aufweist. Als Sonderfall für ein fast zu 100% aus Faltblattstruktur aufgebautes Protein ist das Fibroin zu nennen, wohingegen die meisten Proteine erheblich weniger Faltblattstrukturanteile aufweisen. Bemerkenswert ist die Möglichkeit der reversiblen Umwandlung zwischen α-Helixstruktur und Faltblattstruktur beim Keratin. Für viele Proteine charakteristisch ist die Gruppierung von Sekundärstrukturelementen zu sog. Supersekundärstrukturen (z.B. βαβ). Bereiche, die weder α- noch β-Konformation aufweisen, werden als Zufallsknäuel (auch ungeordnete Gerüstkonformation oder random coil) bezeichnet, die nicht eigentlich zu den Sekundärstrukturen gezählt werden, da sie – abweichend von obengenannter Definition – weder H-Brücken noch repetitive Strukturen enthalten (andere Definitionen: Sekundärstruktur).
Als Tertiärstruktur der Proteine bezeichnet man die Faltung einzelner Peptidketten zu einer dreidimensionalen spezifischen Struktur, wobei die Sekundärstrukturen erhalten bleiben ( vgl. Abb. 1). Durch die Ausbildung von Tertiärstrukturen erhalten Proteine die für ihre jeweiligen Funktionen charakteristische Form (Struktur-Funktion-Beziehung), insbesondere die spezifischen Furchungen der Oberflächen, wie sie z.B. für die Bindung und Umsetzung von Substratmolekülen in den aktiven Zentren der Enzyme erforderlich sind. Bei den zur Ausbildung von Tertiärstrukturen erfolgenden Faltungen gelangen häufig Aminosäurereste, die in den Primär- und Sekundärstrukturen voneinander weit entfernt sind, in räumliche Nachbarschaft. Allosterische Umwandlungen (Allosterie) von Proteinen gehen mit Umfaltungen von Tertiärstrukturen (und als Folge davon auch der Quartärstrukturen, s.u.) einher. Die Stabilisierung von Tertiärstrukturen erfolgt durch intramolekulare (intrachenare) Disulfidbindungen, besonders aber durch die Ausrichtung möglichst vieler hydrophober und unpolarer Aminosäurereste im Innern und hydrophiler, besonders ionisch aufgebauter Aminosäurereste zur äußeren Oberfläche der Strukturen. Dabei fügen sich die hydrophoben Reste der Innenteile meist lückenlos aneinander zu hydrophoben Bereichen, in denen kaum Hohlräume vorhanden sind und die selbst für die relativ kleinen Wassermoleküle (Wasser) des umgebenden Mediums nicht erreichbar sind. Dagegen bilden die polaren Reste der an den Oberflächen gelegenen Aminosäuren Wasserstoffbrücken mit den Wassermolekülen (Wasser) des Mediums aus und führen so zur sog. Hydrathülle der Proteine. Bei Membranproteinen nehmen auch unpolare Aminosäuren einen erheblichen Teil der nach außen gerichteten Positionen ein und ermöglichen so die Verankerung in den hydrophoben Innenbereichen der Membrandoppelschichten (Flüssig-Mosaik-Modell, Membran). Ein weiteres Tertiärstrukturelement bilden sog. Domänen, kompakte Bereiche mit meist eigenständiger Funktion, die oft von eigenen Exonen codiert werden.
Als Quartärstruktur bezeichnet man den Aufbau vieler Proteine aus 2 oder mehreren gleichen oder ungleichen Peptidketten (Untereinheiten, Protomere), die sich nach Ausbildung und unter Erhaltung der Sekundär- und Tertiärstrukturen zu multimeren Proteinen aneinanderlagern ( vgl. Abb. 1 ). Die monomeren Formen sind meist inaktiv. Die Aneinanderlagerung erfolgt normalerweise durch nicht-kovalente Bindungen (ionische Bindungen, hydrophobe Wechselwirkungen, Wasserstoffbrücken), wobei die interagierenden Oberflächenstrukturen der einzelnen Untereinheiten nach dem Schlüssel-Schloß-Prinzip ineinander passen, in seltenen Fällen auch durch Ausbildung von intermolekularen (= interchenaren) Disulfidbrücken. In der Regel besitzen allosterisch regulierbare Proteine eine Quartärstruktur (z.B. Hämoglobin), die durch Bindung eines Effektors veränderbar ist. Allerdings gibt es auch eine Reihe von Proteinen, die aus nur 1 Peptidkette aufgebaut sind (z.B. Lysozym, Myosin, Ribonuclease, bakterielle DNA-Polymerase), denen also eine Quartärstruktur fehlt. Unter den bekannten Proteinen mit Quartärstruktur überwiegen die aus einer geraden Anzahl von gleichen oder ähnlichen Untereinheiten aufgebauten (z.B. Chaperonine, Glutamin-Synthetase, Hämoglobin). Dagegen sind Proteine mit einer ungeraden Zahl oder unterschiedlich großen Untereinheiten (z.B. Immunglobuline) oder mit Untereinheiten, die unabhängig voneinander aktiv sind (Multienzymkomplexe wie z.B. Fettsäure-Synthetase-Komplex, Pyruvat-Dehydrogenase), sowie mit regulatorischen und katalytischen Untereinheiten (Aspartat-Transcarbamylase, cAMP-abhängige Proteinkinase) seltener anzutreffen. Gründe für die Evolution oligomerer Proteinkomplexe (Oligomere) dürften u.a. darin liegen, daß die direkte Interaktion verschiedener funktioneller Bereiche Kooperativität und Rückkopplungsmechanismen (Endprodukthemmung) zwischen den Untereinheiten sowie die Kanalisierung von Metaboliten (metabolic channeling) ermöglicht.
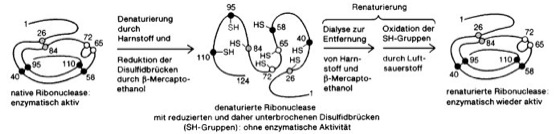
Eigenschaften: Entsprechend ihrer Molekülgröße und -gestalt (Abmessungen 2–100 nm) gehören die Proteine zu den Kolloiden (kolloid). Sie dialysieren (Dialyse) nicht, bilden keine echten Lösungen, zeigen den Tyndall-Effekt und weisen eine relativ hohe Viskosität auf. Proteine tragen eine große Anzahl ionisierbarer Gruppen. Die Ampholytnatur (Ampholyte) der Proteine beruht auf der gleichzeitigen Anwesenheit freier saurer und basischer Gruppen. Sie ist von entscheidender Bedeutung für deren Pufferwirkung (Puffer) in biologischen Systemen. Der Ladungszustand (elektrische Ladung) des Gesamtmoleküls hängt vom pH-Wert des umgebenden Mediums ab ( vgl. Abb. 4 ). Im stark sauren Medium liegen Proteine als Poly-Kationen, im stark basischen als Poly-Anionen vor, deren Überschußladung zunehmende Hydratation und Löslichkeit bedingen. Der Ladungssinn ist für die Wanderungsrichtung im elektrischen Feld verantwortlich. Am isoelektrischen Punkt haben die Proteine keine Nettoladung, Löslichkeit und Hydratation erreichen ein Minimum. Solche Eigenschaften werden bei den verschiedenen Methoden zur Proteinreinigung (vgl. unten) ausgenutzt. Durch Einwirkung bestimmter Agenzien (z.B. Wasserstoffbrücken-lösende Agenzien wie Harnstoff und Guanidiniumhydrochlorid zusammen mit Mercaptoethanol oder Detergentien wie SDS, durch die hydrophobe Wechselwirkungen gelöst werden), aber auch durch nichtphysiologische Bedingungen, wie hohe Temperaturen und pH-Wert-Veränderungen, lösen sich die Überstrukturen (= Summe von Sekundär-, Tertiär- und Quartärstrukturen) der meisten Proteine auf, wobei die Primärstruktur in Form eines Zufallsknäuels (random coil) erhalten bleibt. Diese als denaturiert (Denaturierung) bezeichneten Proteine zeigen gegenüber den nativen Proteinen (nativ) völlig veränderte physikalische und chemische Eigenschaften. Meist sind denaturierte Proteine wasserunlöslich und flocken aus (Fällung), außer sie werden durch Detergentien wie SDS künstlich in Lösung gehalten. Auch die biologische Aktivität, z.B. die katalytische Aktivität von Enzymproteinen, geht durch Denaturierung meist verloren. Die Denaturierung kann bei einfachen Proteinen durch kontrollierte Beendigung der denaturierenden Bedingungen (z.B. durch Dialyse von Harnstoff und Mercaptoethanol) ohne Zuhilfenahme anderer Faktoren rückgängig gemacht werden (Renaturierung; vgl. Abb. 5 ), was als Beweis dafür gilt, daß die Information zur Bildung von dreidimensionalen Strukturen allein in den Aminosäuresequenzen – d.h. letztlich in den Nucleotidsequenzen der betreffenden Gene – enthalten ist. Für die Funktion von Proteinen ist von wesentlicher Bedeutung, daß die Differenz der freien Energie (Enthalpie) zwischen dem entfalteten und dem gefalteten Zustand so gering ist (etwa 40 kJ/mol bei einem Protein mit 100 Aminosäuren), daß der Beitrag des einzelnen Aminosäurerestes zur Stabilität des Proteins im Durchschnitt unter der thermischen Energie (2,5 kJ/mol bei Zimmertemperatur) liegt. Dies erleichtert die Proteinfaltung sowie den Proteintransport durch Membranen und verleiht dem nativen Protein die notwendige Flexibilität für Konformationsänderungen.
Proteinfaltung: Die spontane Faltung der Proteine zu den Überstrukturen – sei es während der Synthese oder durch Renaturierung im Reagenzglas – kann nicht durch Zufallsprozesse unter „Ausprobieren“ aller theoretisch möglichen Faltungskonformationen erfolgen, da dies zu lange dauern würde: Hätte jeder Aminosäurerest nur 3 mögliche Konformationen, deren „Test“ nur 10–13 s beanspruchen würde, so wäre die erforderliche Zeit für ein aus 100 Aminosäuren aufgebautes Peptid 3100 · 10–13 s = 5 · 1047 · 10–13 s = 5 · 1034 s = 1,6 · 1027 Jahre! Da die für die Faltung erforderliche Zeit jedoch im Sekunden- (oder höchstens Minuten-)Bereich liegt, enthalten die Primärstrukturen offensichtlich Informationen für einen möglichst kurzen Faltungsweg zur nativen Endkonformation. Die native Proteinkonformation stellt offenbar auch die thermodynamisch günstigste Konformation dar, was die spontane Faltung der Proteine aus der Primärstruktur nahelegt, wie es auch für einige Proteine in vitro nachvollzogen werden konnte. Ein Grundprinzip dabei ist die fortschreitende Stabilisierung von Zwischenprodukten der Faltung, die der nativen Konformation nahe kommen. Dies geschieht durch Wechselwirkungen, die auch das fertige Protein stabilisieren. Da allerdings der Beitrag des einzelnen Aminosäurerestes zur Stabilität des Proteins sehr gering ist (s.o.), können Zwischenprodukte des Faltungsvorganges verloren gehen. Umgekehrt können Zwischenprodukte, die nicht zu einer nativen Form führen, energetisch günstiger liegen und sog. kinetische Fallen darstellen. Solche Probleme können der Grund dafür sein, daß manche Proteine in vitro und in vivo nicht oder nur schwer zu renaturieren bzw. zu falten sind. Der Faltungsvorgang beginnt mit Bildung kurzer Abschnitte (etwa 8–15 Reste) an α-Helices, β-Faltblättern und β-Schleifen. Die beteiligten Aminosäuren bilden in der Regel autonome Faltungseinheiten, die sich auch als eigenständiges Peptid zu der entsprechenden Struktur falten würden. Diese Faltungseinheiten dienen als Kerne zur Stabilisierung anderer geordneter Regionen, wobei Kerne innerhalb von Millisekunden gebildet und aufgelöst werden können ( vgl. Abb. 6 ). Kerne, die der nativen Konformation angehören, bleiben bestehen und wachsen kooperativ, u.a. durch Bildung von Supersekundärstrukturen und ganzen Domänen. Bei der Faltung von globulären Proteinen tritt eine als „molten globule“ bezeichnete Zwischenstufe mit ausgedehnter Sekundärstruktur, jedoch ungeordneter Tertiärstruktur auf, in der sich hydrophobe Kernbereiche zu einer kompakten, aber noch flexiblen Struktur zusammenlagern und die mit geringen Konformationsänderungen die kompakte Tertiärstruktur des monomeren Proteins erreicht. Im Fall von Proteinen, die durch Disulfidbindungen stabilisiert werden, treten im Verlauf der Faltung nur native Disulfidbindungen auf. Prinzipiell können im Verlauf des Faltungsvorgangs unterschiedliche Regionen zu verschiedenen Zeiten stabilisiert und Faltungswege über verschiedene Zwischenprodukte eingeschlagen werden. Zur Bildung von oligomeren Proteinen lagern sich lediglich die beteiligten Monomere über wenige Zwischenstufen zur nativen Quartärstruktur zusammen (assembly). Problematisch für die Proteinfaltung in vivo sind – neben den bereits genannten Problemen – die relativ hohen Proteinkonzentrationen und Temperaturen in der Zelle, die ständige Wechselwirkungen zwischen Intra- und Interpolypeptidoberflächen zur Folge haben. Dies führt bei der Proteinfaltung im Zusammenhang mit der Proteinsynthese, dem Proteintransport durch Membranen und der Renaturierung nach Streßsituationen sowie beim assembly von Untereinheiten zu Strukturen höherer Ordnung zwangsläufig zu falscher Faltung und Aggregation. Heute weiß man, daß sog. molekulare Chaperone (Polypeptidketten bindende Proteine) bei der Proteinfaltung bzw. beim Übergang von Polypeptidketten aus dem denaturierten in den nativen Zustand sowie beim assembly von Proteinuntereinheiten assistieren (assisted self-assembly). Dabei haben molekulare Chaperone keine klassisch katalytische Wirkung und keinen Einfluß auf die native Konformation, vielmehr verhindern sie hydrophobe Interaktionen und unkorrekte Aggregationen von Intra- und Interpolypeptidoberflächen der Substratproteine. Katalytisch wirksame Faltungshelferproteine sind die Protein-Disulfid-Isomerase zur Bildung korrekter Disulfidbrücken im nicht reduzierenden Milieu des endoplasmatischen Reticulums und die Peptidyl-Prolyl-cis-trans-Isomerase zur cis/trans-Isomerisierung von Prolinpeptidbindungen.
Einteilung: Die Einteilung der Proteine kann nach mehreren Kriterien wie Zusammensetzung und Form ( vgl. Tab. ), Vorkommen und Funktion erfolgen. – Nach der Zusammensetzung unterscheidet man einfache Proteine, die nur aus proteinogenen Aminosäuren aufgebaut sind, und zusammengesetzte Proteine (konjugierte Proteine), die neben dem Proteinanteil eine meist chemisch gebundene Nichtproteinkomponente enthalten. – Nach der Form unterscheidet man globuläre und fibrilläre Proteine. Globuläre Proteine (Sphäroproteine) sind kugelförmig und in Wasser und verdünnten Salzlösungen löslich. Dies beruht auf den an der Moleküloberfläche lokalisierten, geladenen, hydrophilen Aminosäureresten, die – umgeben von einer Hydrathülle – für einen engen Kontakt mit dem Lösungsmittel sorgen. Die fibrillären Proteine (Faserproteine, Strukturproteine, Skleroproteine) sind in Wasser und Salzlösungen praktisch unlöslich. Die Polypeptidketten sind hier parallel zueinander geordnet und bilden lange Fasern. – Nach dem Vorkommen in den Organismengruppen ist zwischen menschlichen, tierischen, pflanzlichen und mikrobiellen Proteinen zu unterscheiden, wobei weitere Unterteilungen nach dem Vorkommen in bestimmten Organen (z.B. Blutproteine, Serumproteine, Milchproteine, Muskelproteine, Vorratsproteine pflanzlicher Samen) und in bestimmten Zellfraktionen (Cytoplasmaproteine, Ribosomenproteine, Kernproteine, Membranproteine, Chloroplastenproteine und Mitochondrienproteine) oder Viren (Capsid) gemacht werden. – Nach der Funktion unterteilen sich die Proteine in: 1) Enzymproteine (Enzyme), welche die biochemischen Reaktionen des Stoffwechsels katalysieren. 2) Transportproteine für den Transport von Sauerstoff, Fettsäuren, Hormonen, Metallionen und Elektronen (z.B. Hämoglobin, Myoglobin, Transferrin, Coeruloplasmin, Serumalbumin, Cytochrome) oder Translokatorproteine des Membrantransports (Translokator). 3) Speicherproteine für die Speicherung von Metallionen (z.B. Hämosiderin, Ferritin) oder Vorratsproteine, welche die Aminosäurereserve des Organismus sichern (z.B. Eieralbumin, Casein, Gliadin, Gluteline, Prolamine, Zein). 4) Kontraktile Proteine, welche die koordinierte Beweglichkeit bestimmter Organe (z.B. Muskel; Muskelproteine), Organelle (z.B. Cilien) oder sonstiger zellulärer Strukturen (z.B. Chromatiden) verursachen (z.B. Actin, Myosin, Mikrotubuli, Dynein, Kinesin; Motorproteine). 5) Struktur- bzw. Gerüstproteine (Skleroproteine) als wesentlicher Bestandteil von Stützgewebe und Bindegewebe (z.B. Kollagen, Elastin, Keratine). 6) Abwehrproteine für zelluläre Abwehrreaktionen (z.B. Immunglobuline). 7) Rezeptorproteine für die Weiterleitung von durch Hormone oder Neurotransmitter bedingten Signalen. 8) Regulatorproteine, die an regulatorischen Prozessen der Genaktivierung (Genregulation) und des Zellwachstums beteiligt sind (z.B. Aktivatorproteine und Repressorproteine [Repressoren], Proteohormone, Transkriptionsfaktoren, Wachstumsfaktoren). 9) Zelloberflächenproteine, die die Erkennung eines Zelltyps durch einen anderen ermöglichen und deshalb eine Rolle bei der Morphogenese und der Erkennung von fremdem Gewebe spielen (z.B. Adhäsine, Blutgruppenantigene, CD-Marker). 10) Faktoren der Blutgerinnung und Fibrinolyse. Diese Vielzahl von hochspezifischen Funktionen ist in Anbetracht der Strukturvielfalt der Proteine verständlich. Aus ihr resultiert auch der mengenmäßig hohe Anteil der organischen Bestandteile an Protein (über 50%), den die Zellen aller Organismen (auch die Viren) aufweisen. –
Wichtige Techniken und Parameter zur Charakterisierung von Proteinen:
a) Quantitative Bestimmung und Nachweisverfahren: Diese beruhen entweder auf Farbreaktionen (Kolorimetrie, Proteinbestimmung), die für bestimmte Aminosäurereste spezifisch sind, auf der Messung der Absorption (Absorptionsspektroskopie, Extinktion) bei 280 nm Wellenlänge, die durch die aromatischen Aminosäurereste bedingt ist, oder auf Messung der Absorption von sichtbarem Licht bei Chromoproteinen. Bei Farbreaktionen werden zur Standardisierung Lösungen von Eichproteinen (meist Rinderserumalbumin) bekannter Konzentration in Parallelansätzen zur Reaktion gebracht und durch Vergleich der Farbintensitäten die Konzentrationen der zu untersuchenden Proteine bestimmt. Diese Verfahren sind für die meisten Proteine anwendbar, erlauben jedoch keine Unterscheidung zwischen einzelnen Proteinen oder Proteingemischen. Dagegen eignen sich immunologische Verfahren (Immunassays), z.B. die Immunpräzipitation (Agardiffusionstest, Immunelektrophorese) oder die Western-Technik (blotting-Techniken), als besonders spezifische und empfindliche Nachweismethoden für einzelne Proteine in komplexen Gemischen, wie Blutseren (Serumproteine) und Zellaufschlüssen. Die hohe Spezifität dieser Nachweismethoden beruht auf der Wirkung von Proteinen als Antigene bzw ihrer Bindung an Antikörper (Immunglobuline). Durch den Einbau radioaktiver Gruppen durch chemische Methoden oder radioaktiver Aminosäuren während der Proteinsynthese markierte (Markierung) Proteine sind z.B. mittels Autoradiographie und Radioimmunassays detektierbar. Enzymproteine können außerdem durch die von ihnen katalysierten Reaktionen spezifisch nachgewiesen werden, wobei die gängigste Methode der optische Test ist.
b) Reinigung und Isolierung: Lösliche intrazelluläre Proteine werden präparativ aus den meist sehr komplexen Gemischen, wie sie z.B. in Homogenaten nach Zellaufschluß vorliegen, gereinigt. Bei extrazellulären Proteinen, z.B. aus Körperflüssigkeiten oder Überständen von Zellkulturen, müssen zunächst Zellen und andere unlösliche Bestandteile entfernt werden. Nichtlösliche Proteine, wie z.B. die meisten integralen Membranproteine, müssen vor Beginn eines Reinigungsverfahrens durch Detergentien wie CHAPS oder SDS solubilisiert oder durch chaotrope Verbindungen an der Ausbildung hydrophober Interaktionen gehindert werden, wobei die Erhaltung des nativen Zustands meist nicht mehr möglich ist. Bei den ebenfalls unlöslichen Strukturproteinen (Skleroproteine) ist es in der Regel sinnvoll, vor der Anreicherung zuerst alle löslichen Proteine zu entfernen. Rekombinante Proteine sind recht einfach zu reinigen, da sie in relativ großen Mengen verfügbar sind und die Reinigungsstrategie auf die Art ihrer Expression (z.B. Fusionsproteine mit spezifischen Tags) und Freisetzung (z.B. in Form von inclusion bodies [Einschlußkörperchen], Sezernierung in Kulturmedium) abgestimmt werden kann (Expressionssystem). Der eigentliche Reinigungsvorgang der Proteine erfolgt unter Ausnutzung ihrer Größe, Löslichkeit, Ladung, spezifischen Bindungseigenschaften und biologischen Aktivität durch Kombinationen verschiedener Methoden: Mittels fraktionierter Zentrifugation (Dichtegradienten-Zentrifugation und differentielle Zentrifugation) und fraktionierter Fällung (ausfällen), z.B. mit Ammoniumsulfat, Ethanol (Cohn-Fraktionierung) oder Trichloressigsäure, wird eine Vortrennung durchgeführt. Weitere Reinigungsschritte basieren auf chromatographischen (Chromatographie) und elektrophoretischen (Elektrophorese) Trennmethoden, u.a. Ionen-Austauschchromatographie, Adsorptionschromatographie, Affinitätschromatographie, Metallchelat-Affinitätschromatographie, Reversed-Phase-Chromatographie, Hydrophobe Interaktionschromatographie, Gelfiltration, präparative Gelelektrophorese und isoelektrische Fokussierung. Grundsätzlich ist die Strategie der einzelnen Reinigungsschritte, entweder unerwünschte Komponenten zu entfernen oder die Zielkomponente anzureichern. Zwischen den einzelnen Reinigungsschritten ist es häufig notwendig, die Ionenstärke oder Pufferzusammensetzung der Lösungen anzupassen, was z.B. mittels Dialyse, Ultrafiltration oder Gelfiltration erreicht werden kann. Eine Konzentrierung von Lösungen ist ebenfalls durch Dialyse und Ultrafiltration sowie durch Fällung möglich. Die Entfernung von Detergentien bei der Reinigung hydrophober Proteine (wodurch deren Löslichkeit und Aktivität verloren gehen) kann u.a. durch Extraktion mit Chloroform/Methanol oder durch Reversed-Phase-Chromatographie erfolgen. Ein zur Homogenität (Einheitlichkeit) gereinigtes Protein ist für die nachfolgende Analytik von Struktur und Funktion von wesentlicher Bedeutung.
c) Reinheitskriterien: Ein „reines“ Protein kann noch anorganische Salze, andere kleine organische Moleküle (z.B. Substrate, Coenzyme) und Wasser enthalten. Der Reinheitsgrad von Proteinen bzw. die Zusammensetzung von Proteingemischen wird meist durch Gelelektrophorese ermittelt, wobei entweder eindimensionale Verfahren, wie Disk-Elektrophorese, isoelektrische Fokussierung und SDS-Polyacrylamid-Gelelektrophorese, angewandt werden oder zur Auflösung sehr komplexer Proteingemische (bis über 1000 verschiedene Proteine) die hochauflösende zweidimensionale Gelelektrophorese eingesetzt wird. Kriterien für die Reinheit von Proteinen sind ferner einheitliche Sedimentationsgeschwindigkeit (S-Wert; Sedimentation) bei Ultrazentrifugation (Ultrazentrifuge), Ganzzahligkeit der Aminosäurezusammensetzung, Sequenzierbarkeit (Sequenzierung), linearer Verlauf bis zum Sättigungspunkt im Löslichkeitsdiagramm und Kristallisierbarkeit (Proteinkristallisation). Bei Enzymen kommen die Aktivitätskriterien (pH- und Temperatur-Optimum, Substratspezifität, kinetisches Verhalten) als weitere Reinheitskriterien hinzu.
d) Relative Molekülmassen („Molekulargewichte“) und Kettenlängen: Diese können zumindest annäherungsweise unter gleichzeitiger Denaturierung durch Vergleich der Laufgeschwindigkeiten der zu untersuchenden Peptidketten mit den Laufgeschwindigkeiten von Markerproteinen (Marker) von bekannter Molekülmasse in der SDS-Polyacrylamid-Gelelektrophorese bestimmt werden. Die Molekülmassen nativer Proteine lassen sich aus den Sedimentationsgeschwindigkeiten bei Ultrazentrifugation (insbesondere bei Dichtegradienten-Zentrifugation) und dem Elutionsvolumen bei der Gelfiltration abschätzen. Aus den Molekülmassen können die Kettenlängen (= Anzahl von Aminosäureresten pro Peptidkette) annähernd errechnet werden, indem man sie durch die Zahl 110, die mittlere Molekülmasse der 20 Aminosäurereste, dividiert. Sind die Aminosäurezusammensetzung und die Anzahl der tryptischen Peptide oder die Aminosäuresequenz eines Proteins bekannt, kann umgekehrt die Molekülmasse rechnerisch ermittelt werden. Verschiedene Verfahren der Massenspektrometrie erlauben inzwischen die Bestimmung der relativen Molekülmassen kleinster Proteinmengen mit hoher Massenauflösung und einem hohen Probendurchsatz, was im Rahmen der Proteomik von wesentlicher Bedeutung ist.
e) Anzahl von Peptidketten (und deren Molekülmassen): Diese sind nach Denaturierung durch SDS-Polyacrylamid-Gelelektrophorese direkt bestimmbar, sofern verschieden lange und/oder verschieden zusammengesetzte Ketten am Aufbau eines gereinigten Proteins beteiligt sind. Bei oligomer aus identischen Ketten aufgebauten Proteinen kann die Kettenzahl durch Vergleich der Molekülmasse des nativen Proteins (bestimmbar aus der Sedimentationsgeschwindigkeit) mit der Molekülmasse des denaturierten Proteins (bestimmbar aus SDS-Polyacrylamid-Gelelektrophorese) ermittelt werden. Bei einem aus 4 gleichen Untereinheiten aufgebauten Protein unterscheiden sich diese Molekülmassen z.B. durch den Faktor 4.
f) Aminosäurezusammensetzung: Diese wird nach Hydrolyse des Proteins zu den Aminosäuren im Aminosäureanalysator bestimmt.
g) Aminosäuresequenz: Zur direkten Ermittlung der Aminosäuresequenz eines Proteins wird dieses zunächst – nach Spaltung der Disulfidbindungen – durch limitierte Einwirkung von Proteasen oder durch die Bromcyan-Reaktion in definierte Teilpeptide gespalten und diese chromatographisch getrennt. Anschließend werden die Aminosäuresequenzen aller Teilpeptide einzeln durch die jeweils am Aminoterminus beginnenden zyklischen Reaktionsfolgen des Edmanschen Abbaus (z.B. im Gasphasen-Sequenator) und/oder durch Massenspektrometrie ermittelt. (C-terminale Endgruppen werden separat durch chemische oder enzymatische Abbaumethoden oder massenspektrometrisch bestimmt.) Durch eine Serie von überlappenden Teilpeptiden kann schließlich die Aminosäuresequenz der gesamten Kette abgeleitet werden. In der Regel werden heute Aminosäuresequenzen von Proteinen aus den Nucleotidsequenzen der betreffenden Gene nach den Regeln des genetischen Codes abgeleitet, denn mit der rasanten Entwicklung und Erweiterung von Methoden im Rahmen der Gentechnologie und des Genomprojekts ist die Sequenzanalyse von DNA technisch einfacher als die von Proteinen (DNA-Sequenzer; Sequenzierung). Die direkte Aminosäuresequenzierung beschränkt sich auf leicht zugängliche oder ausgewählte Teilpeptide zur stichprobenartigen Kontrolle der von Nucleotidsequenzen abgeleiteten Aminosäuresequenzen bzw. zur Bestimmung von nicht ableitbaren Aminosäuresequenzen, wie z.B. der Exon-Grenzen (Exon, Mosaikgene).
h) Strukturanalyse: Die Sekundärstrukturen von Proteinen lassen sich z.B. mittels CD-Spektroskopie und Infrarotspektroskopie erfassen. Tertiärstrukturen – einhergehend damit oft auch die Sekundär- und Quartärstrukturen – werden in der Regel durch Röntgenstrukturanalyse an isomorph kristallisierten Schwermetallatomderivaten des betreffenden Proteins ermittelt, während durch Messung des Eigendrehimpulses (Spin) von Atomkernen mit Hilfe der Kernresonanzspektroskopie zusätzlich zur Proteinstrukturbestimmung (in Lösung) die Erfassung dynamischer Phänomene an Proteinen möglich ist. Quartärstrukturen von Proteinen können durch elektronenmikroskopische Verfahren (Elektronenmikroskop) inklusive verschiedener Verfahren zur Bildanalyse, Bildverarbeitung und 3D-Rekonstruktion (bildgebende Verfahren) analysiert werden. Methoden mit dem Schwerpunkt Struktur-Funktions-Analyse sind – neben fast allen spektroskopischen Verfahren – u.a. die chemische Modifikation funktioneller Gruppen, die Einführung von Reportergruppen (z.B. Fluorochrome; Fluoreszenzmarkierung, Fluoreszenzspektroskopie), das Protein-Cross-link, die Peptidsynthese und die gezielte Veränderung der Aminosäuresequenz durch in-vitro-Mutagenese des codierenden Gens (protein engineering). Häufig ist es außerdem notwendig, ausreichende Mengen eines Proteins für die Analyse mittels gentechnologischer Methoden (Gentechnologie) zu gewinnen. –
Zur historischen Entwicklung der Proteinforschung: Biochemie (Geschichte der); zur Synthese von Proteinen: Translation, endoplasmatisches Reticulum, Exportproteinsynthese; zum Abbau der Proteine: Proteasen, Proteinabbau, Proteolyse.
Bedeutende Forschungsarbeiten auf dem Gebiet der Proteine leisteten u.a. C.B. Anfinsen, C.G.R. Berg, C.L. von Berthollet, T.R. Cech, J. Deisenhofer, Em.H. Fischer, H. Fraenkel-Conrat, A.D. Hershey, E.F.I. Hoppe-Seyler, R. Huber, J.C. Kendrew, A. Klug, A. Kossel, O. Loewi, F. Magendie, R.B. Merrifield, H. Michel, G.E. Palade, L.C. Pauling, M.F. Perutz, W. Prout, F. Sanger, J.B. Sumner. – Bioinformatik, Biophysik, Brennwert, Ernährungsphysiologie (Tab.), essentielle Nahrungsbestandteile, Genomik, Kerntransport von Proteinen, Molekularbiologie, Nahrungsmittel, Nahrungsstoffe, Nährwert, Prionen, Proteinbestimmung, Protein-DNA-Interaktion, Protein-Energie-Mangelsyndrom, Proteinfamilie, Proteinkristallisation, Protein-Liganden-Komplex, Protein-Protein-Interaktionen, Proteinsekretion, Protein-Sequenator, Protein-Signatur, Protein-sorting, Protein-Spleißen, Proteinstoffwechsel, Protein-targeting, Protein-Werkstoffe, Proteom, Proteomik, Ribosomen (Farbtafel), Röntgenstrukturanalyse, spleißen, Stoffwechsel (Farbtafel), Strukturvorhersage, Zellwandproteine; Proteine I Proteine II , ä chemische und präbiologische Evolution , Transkription–Translation.
H.K./P.Z./M.B.
Lit.: Branden, C., Tooze, J.: Introduction to protein structure. New York 1991. Kyte, J.: Structure in protein chemistry. New York 1995. Lehninger, A.L., Nelson, D.L., Cox, M.M.: Prinzipien der Biochemie. Heidelberg 21996. Stryer, L.: Biochemie. Heidelberg 41996.
Proteine
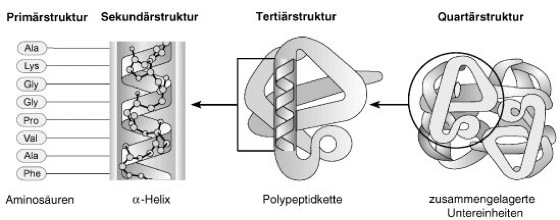
Abb. 1: Strukturebenen von Proteinen:
Die Primärstruktur entspricht der Sequenz der durch Peptidbindungen miteinander verbundenen Aminosäuren. Die entstehenden Polypeptide können, wie in vorliegendem Beispiel, zu einer α-Helix gewunden sein, die eine der möglichen Sekundärstrukturen darstellt. Die Helix ist Teil der Tertiärstruktur des gefalteten Polypeptids. Dieses wiederum stellt eine der Untereinheiten dar, welche die Quartärstruktur des multimeren Proteins, im vorliegenden Beispiel des Hämoglobins, bilden.
Proteine
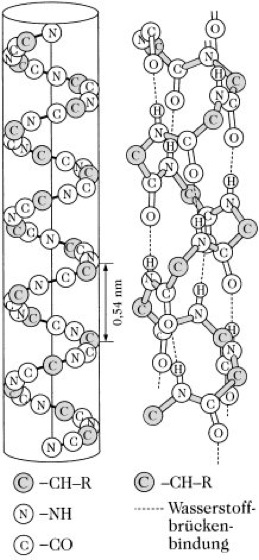
Abb. 2: α-Helixstruktur: α-Helix (rechtsgängig) mit ihren Wasserstoffbrücken-Bindungen, links in idealisierter (vereinfachter) Darstellung zur Veranschaulichung des „Bauprinzips“. Maße der α-Helix: Anstieg pro Aminosäureeinheit 0,15 nm, Ganghöhe 0,54 nm = 3,6 Aminosäuren, Durchmesser etwa 2 nm, abhängig von der Größe der Seitenketten (R)
Proteine
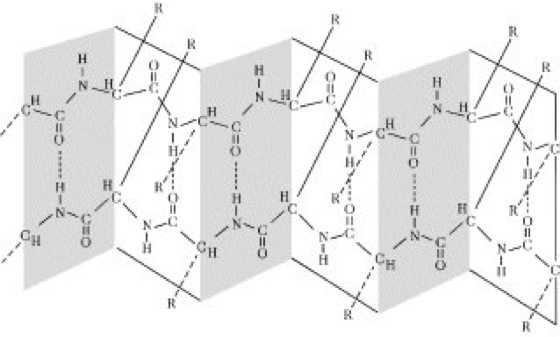
Abb. 3: Faltblattstruktur (β-Struktur). Die Aminosäureseitengruppen (R) stehen abwechselnd oberhalb und unterhalb der Blattstruktur. Allzu große Seitengruppen behindern die Ausbildung der Faltblattstruktur.
Proteine
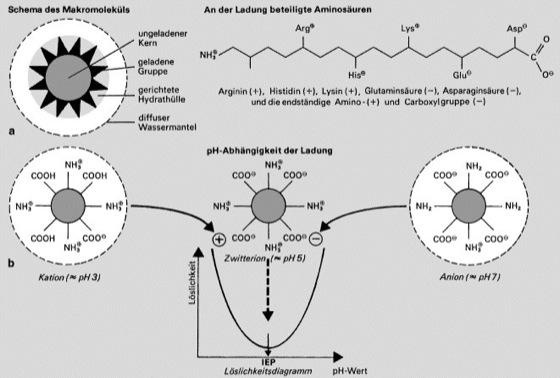
Abb. 4: a Schematischer Aufbau globulärer Proteine aus ungeladenem Kern („Öltröpfchen“), geladenen Gruppen in der Peripherie, Hydrathülle und Wassermantel. b Durch den Aufbau aus positiv und negativ geladenen Aminosäuren sind Proteine Zwitterionen. Als isoelektrischer Punkt (IEP) ist derjenige pH-Wert definiert, bei dem ein Protein gleichviel positive wie negative Ladungen aufweist; bei diesem pH-Wert zeigt die Löslichkeit ein Minimum. Durch Verschiebung des pH-Werts zur sauren (bzw. alkalischen) Seite des isoelektrischen Punkts werden negative (bzw. positive) Ladungen neutralisiert, wodurch die Gesamtladung zunehmend positiv (bzw. negativ) wird, d.h. das betreffende Protein zum Kation (bzw. Anion) wird.
Proteine
Abb. 5: Denaturierung und Renaturierung des Enzymproteins Ribonuclease
Proteine
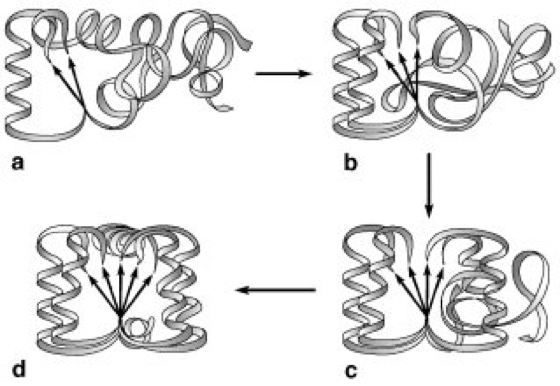
Abb. 6: Eine mögliche Variante der Proteinfaltung:
a Bildung eines strukturellen Kerns, der aus wenigen, besonders stabilen Sekundärstruktur-Bereichen besteht. b Während andere Regionen ihre Sekundärstruktur einnehmen, werden sie über weit entfernte Bereiche durch Wechselwirkungen mit dem strukturellen Kern stabilisiert. c Der Faltungsprozeß setzt sich fort, bis der Großteil des Polypeptids seine korrekte Sekundärstruktur angenommen hat. d Die endgültige Struktur hat normalerweise die thermodynamisch stabilste Konformation.
Schreiben Sie uns!