Lexikon der Biologie: Ribosomen
Ribosomen [von *ribo- , griech. sōma = Körper; Adj. ribosomal], die größten und am kompliziertesten aufgebauten, gleichzeitig stabilsten und zahlreichsten Ribonucleoprotein-Partikel (Abk. RNP-Partikel; Ribonucleinsäuren, Ribonucleoproteine) der Zelle, an denen die Translation der genetischen Information, d.h. die Proteinsynthese, stattfindet. Sie sind im Elektronenmikroskop als rundliche bis ellipsoide Partikel von 15 bis 30 nm Durchmesser erkennbar ( vgl. Abb. 1 ). Während des Translationsprozesses sind gleichzeitig mehrere Ribosomen an jeder mRNA-Kette (messenger-RNA) gebunden und bilden so die Polyribosomen. Erstmals beschrieben wurden Ribosomen 1953 von G.E. Palade als Komponenten des rauhen endoplasmatischen Reticulums (sog. Palade-Granula oder Palade-Körner). Schon kurz darauf gelang die Isolierung freier Ribosomen aus dem Grundplasma (sog. Plasmaribosomen) eukaryotischer Zellen (Eucyte) und aus Bakterienzellen. Dabei zeigte sich, daß man prinzipiell 2 Typen von Ribosomen unterscheiden kann ( vgl. Tab. ): den eukaryotischen 80S-Typ und den prokaryotischen 70S-Typ, dem auch die Mitoribosomen und Plastoribosomen angehören (S = Svedberg-Konstante, Sedimentationskoeffizient; Sedimentation). Auch die Fähigkeit der Ribosomen zur Proteinsynthese (Proteine) wurde noch in den 1950er Jahren erkannt. In den 1960er Jahren wurden die in den Plastiden (Chloroplasten) bzw. Mitochondrien enthaltenen Ribosomen (Plastoribosomen bzw. Mitoribosomen) entdeckt. Eine typische Bakterienzelle (Bakterien, Cyanobakterien) enthält einige 104 Ribosomen, die etwa 1/4 der Zellfrischmasse ausmachen. In eukaryotischen Zellen ist ihre Zahl entsprechend höher (z.B. in Leberparenchymzellen 4·106, in Reticulocyten 3·104), der Massenanteil jedoch geringer. Extrem hohe Werte finden sich in Oocyten und Eizellen (z.B. 1012 in Amphibieneiern; Amphibienoocyte, Genamplifikation). Andererseits sind Zellen ohne aktive Kerne, wie z.B. reife Erythrocyten, Spermien und Siebzellen, frei von Ribosomen. Ribosomen dissoziieren während des Ribosomen-Zyklus der Translation oder künstlich durch Entzug von Mg2+-Ionen (Magnesium) in 2 verschieden zusammengesetzte und verschieden große ribosomale Untereinheiten ( vgl. Abb. 2 ). Aus bakteriellen (prokaryotischen) 70S-Ribosomen entstehen so die 50S-Untereinheiten (große Untereinheiten) und die 30S-Untereinheiten (kleine Untereinheiten). Aus eukaryotischen 80S-Ribosomen bilden sich 60S- und 40S-Untereinheiten. Wie besonders an Ribosomen von Escherichia coli gezeigt wurde, können ribosomale Untereinheiten im Reagenzglas durch Behandlung mit bestimmten Salzen, Detergentien, denaturierenden Agentien (Harnstoff, Phenol; Denaturierung) in freie ribosomale RNAs (Abk. rRNAs) und in die einzelnen Proteine (ribosomale Proteine, Abk. r-Proteine) zerlegt werden, wobei auch Zwischenstufen isolierbar sind. Umgekehrt konnten intakte, d.h. in in vitro-Translationssystemen aktive Ribosomen durch in vitro-Rekonstitution aus isolierten rRNAs und r-Proteinen außerhalb der lebenden Zelle gewonnen werden. Die dabei entstehenden Bindungen der einzelnen r-Proteine an definierten Bereichen der jeweiligen rRNAs bzw. an andere bereits gebundene r-Proteine bilden sich in einer bestimmten Reihenfolge (assembly pathway) aus, wie durch Isolierung bestimmter Teilkomplexe gezeigt werden konnte. Man nimmt an, daß diese in vitro beobachtete Reihenfolge auch bei der Entstehung der Ribosomen in lebenden Zellen durchlaufen wird. Da die Bildung von Ribosomen aus den zahlreichen Protein- und RNA-Komponenten spontan und ohne Einwirkung zusätzlicher Faktoren erfolgt, stellt sie ein Schulbeispiel für die Selbstorganisation (assembly) einer komplexen biologischen Struktur dar ( vgl. Abb. 3 ). – Ribosomale RNAs machen etwa 80% der zellulären Gesamt-RNA aus und bilden zusammen mit tRNAs (ca. 15%; transfer-RNA) – und im Gegensatz zu den weniger stabilen mRNAs (ca. 5%; messenger-RNA) – die sog. stabile RNA-Fraktion. Die Nucleotidsequenzen zahlreicher rRNAs konnten analysiert werden, wobei diejenigen der kleinen rRNAs (5S, 4,5S, 5,8S) durch direkte RNA-Sequenzanalyse, diejenigen der großen rRNAs (16S, 18S, 23S, 26S) fast ausschließlich über die DNA-Sequenzanalyse der entsprechenden Gene (rRNA-Gene) ermittelt wurden. Aufgrund des Vorkommens von rRNA in allen Organismen und der Möglichkeit, Nucleotidsequenzen als quantitative Parameter rechnerisch auszuwerten, eignen sich die Nucleotidsequenzen von rRNAs (besonders von 16–18S-rRNA, aber auch von 5S-rRNA) hervorragend zur Ermittlung phylogenetischer Beziehungen (molekulare Systematik, Sequenzstammbaum). Beispielsweise zeigen die 16S-rRNA-Sequenzen aus Escherichia coli und Chloroplasten von Mais in 74% der ca. 1500 Positionen identische Nucleotide, was den prokaryotischen Charakter der Plastoribosomen und damit die Endosymbiontenhypothese (Evolution der Eucyte) stützt. – Ribosomale RNAs falten sich durch intramolekulare Basenpaarungen zu Sekundärstrukturen (für Escherichia coli 5S und 16S; vgl. Abb. 4 ), die trotz Abweichungen in Einzelelementen und trotz teilweise verschiedener Primärstrukturen eine für alle Organismen gemeinsame Grundstruktur aufweisen. Die weitere Faltung zu einer Tertiärstruktur (analog der L-Struktur von transfer-RNA) ist zur Zeit noch wenig verstanden, wenngleich die Lokalisation einzelner rRNA-Domänen aufgrund ihrer Bindung an r-Proteine und deren Lokalisation im dreidimensionalen Modell ( vgl. Abb. 5 , vgl. Abb. 6 , Ribosomen ) in Ansätzen möglich ist. – Die Synthese von bakterieller und plastidärer rRNA erfolgt durch Transkription der meist in mehreren rRNA-Operonen zusammengefaßten rRNA-Gene. Das Escherichia-coli-Genom enthält 7 rRNA-Operonen, das Genom von Bacillus subtilis 10. Plastiden-DNA Höherer Pflanzen (mit Ausnahme bestimmter Hülsenfrüchtler) enthält 2 rRNA-Operonen. In einer als Primärtranskript zunächst entstehenden Vorläufer-rRNA (rRNA-Präkursor) sind innerhalb einer Kette (sog. 30S-Prä-rRNA bei Escherichia-coli) die kompletten Sequenzen von 16S-, 23S- und 5S- (bei Plastiden auch 4,5S-)rRNA sowie intergenische Bereiche, wie u.a. der 16S/23-spacer mit tRNA-Sequenzen (sog. spacer-tRNAs), enthalten. Durch Prozessierung wird die Vorläufer-rRNA über bestimmte Zwischenstufen (25S-rRNA → 23Sr-RNA; 17S-rRNA -› 16S-rRNA) zu den verschiedenen rRNAs und tRNAs gespalten, wobei gleichzeitig die Modifikation einzelner Basen (ca. 1%; Basenmodifikation) und die Bindung an r-Proteine erfolgt. Die Synthese und Prozessierung von eukaryotischer rRNA vollzieht sich im Nucleolus ebenfalls über eine Vorläufer-rRNA (45S-rRNA bei Säugern), welche die kompletten Sequenzen von 18S-, 5,8S- und 28S-rRNA, nicht jedoch von 5S-rRNA, enthält. 5S-rRNA wird von Genen des Zellkerns außerhalb des Nucleolus (bzw. Nucleolus-Organisators) codiert. Die Vorläufer-rRNA wird schon während ihrer Synthese mit Proteinen beladen, von denen r-Proteine zunächst nur einen kleinen Teil ausmachen. Die entstehenden 80S-rRNP-Partikel sind Ausgangsprodukte für die anschließende Prozessierung der rRNA-Vorläufer zu 18S-, 5,8S- und 28S-rRNA und für die Einschleusung von 5S-rRNA, wobei gleichzeitig auch einzelne Basen modifiziert (bis 1%) bzw. einzelne Ribosereste 2'-O-methyliert (bis 2%) werden und die zunächst gebundenen, nicht-ribosomalen Proteine zunehmend durch r-Proteine ersetzt werden. Diese müssen nach ihrer Synthese an den bereits vorhandenen cytoplasmatischen Ribosomen in den Kern bzw. Nucleolus geschleust werden. Umgekehrt werden die so entstehenden Ribosomen-Vorläufer, die sog. Präribosomen, nach ihrer Bildung im Nucleolus in das Cytoplasma transportiert, wobei gleichzeitig in abschließenden Reifungsschritten die Ausstattung mit r-Proteinen komplettiert wird und einige r-Proteine phosphoryliert (Phosphorylierung) werden. Anschließend können die reifen Untereinheiten in den Ribosomen-Zyklus der Translation eintreten. – Ribosomen sind Multienzymkomplexe, welche die Einzelschritte der Translation fließbandartig katalysieren. In Prokaryoten beginnt der Start der Translation immer mit einer freien 30S-Ribosomenuntereinheit. Dabei bildet sich ein Initiationskomplex aus dieser Untereinheit, N-Formyl-Methionyl-tRNA und Initiationsfaktoren. Die 50S-Untereinheit wird hinzugefügt, so daß ein aktives 70S-Ribosom entsteht. Eine spezielle Sequenz der bakteriellen mRNA, die sog. Shine-Dalgarno-Sequenz, dient als Ribosomenbindungsstelle und ermöglicht das Ablesen polycistronischer mRNA (polycistronische RNA). Eukaryotische Ribosomen erkennen in der Regel eine mRNA an ihrer 5`-Cap-Struktur (Capping) und starten nur am ersten möglichen Start-Codon. Daher kann normalerweise keine polycistronische mRNA übersetzt werden. An der Ribosomen-Oberfläche bzw. in deren Furchen existieren Bindestellen für mRNA (an der kleinen Untereinheit), Aminoacyl-tRNA (A-Bindungsstelle) und Peptidyl-tRNA (P-Bindungsstelle) sowie das Peptidyl-Transferase-Zentrum (an der großen Untereinheit). Durch letzteres wird die Schlüsselreaktion des Ribosoms, der Peptidtransfer von Peptidyl-tRNA auf Aminoacyl-tRNA, katalysiert (Peptidyl-Transferase). Früher wurde angenommen, daß die Translokation der tRNA von Position A zu Position P die Freisetzung der leeren tRNA herbeiführt. Es scheint jedoch so zu sein, daß diese leere tRNA an einen 3. Ort, die sog. Position E (engl. exit), geschoben wird. Erst aus dieser Position wird die tRNA „entlassen“. Das Ribosom muß als dynamische Struktur verstanden werden. Sowohl die mRNA als auch die tRNA bewegen sich in jedem Stadium der Proteinsynthese durch das Ribosom. Die 3 Positionen befinden sich nicht statisch an der Oberfläche, sondern müssen als bewegliche Teile einer komplexen „biomolekularen Maschinerie“ angesehen werden. – Ziel moderner struktureller Untersuchungen ist es, ein räumliches Modell des Ribosoms mit atomarer Auflösung zu gewinnen. Bisher standen hier Vernetzungsstudien, Neutronenbeugungsuntersuchungen, Röntgenstrukturanalyse (insbesondere unter Einsatz von Synchrotronstrahlung; European Synchrotron Radiation Facility) und die Elektronenmikroskopie (Elektronenmikroskop) im Vordergrund. Aus diesen Untersuchungen ergibt sich folgendes Bild: die beiden ribosomalen Untereinheiten bilden einen internen Raum für den Ablauf der Translation. Die mRNA tritt vermutlich durch einen Kanal in der kleinen Untereinheit ein und verläßt den Raum seitlich nach einer kurzen Schleife. Der Ort der Interaktion von mRNA und tRNA ist auf eine Region von etwa 2 nm begrenzt. Die wachsende Polypeptidkette verläßt den internen Raum durch einen ca. 8 nm langen Tunnel durch die große Untereinheit, der etwa 40 Aminosäureresten Platz bietet. Mit Hilfe der Röntgenstrukturanalyse konnte zwischen 1999 und 2001 die Kristallstruktur der großen 50S-Untereinheit des Archaebakteriums Haloarcula marismortui mit einer atomaren Auflösung von 2,9 Å sowie die Struktur der kleinen ribosomalen Untereinheit von Thermus thermophilus mit einer Auflösung von 3,3 Å dargestellt werden ( Ribosomen ). Dabei bestätigte sich eine lange Zeit gehegte Vermutung: Die funktionellen Zentren der Ribosomen bestehen größtenteils aus rRNA. Die Peptidyl-Transferase-Reaktion wird durch einen Abschnitt der 23S-rRNA katalysiert. Die wichtigen funktionellen Bereiche der kleinen Untereinheit, das Decodierungszentrum und der Eingang des mRNA-Tunnels, werden ebenfalls von ribosomaler RNA gebildet. Diese ribosomale RNA ist z.B. für die Organisation des Codon-Anticodon-Zusammenspiels verantwortlich. Die ribosomalen Proteine sind eher als Stütze des RNA-Skeletts sowie für die Stabilisierung des gesamten Partikels zuständig. Vermutlich dirigieren sie auch Co-Faktoren wie tRNA und Initiationsfaktoren an die richtige Stelle. Das Ribosom ist also ein Ribozym, d.h., die ribosomale RNA weist, vergleichbar den Proteinenzymen, ein aktives Zentrum auf, an das Substrate binden, und katalysiert die Bildung eines Produkts. – Große und kleine Untereinheit prokaryotischer Ribosomen enthalten je nach Spezies 30–40 bzw. etwa 20 ribosomale Proteine, die als L1–L40 (large subunit) oder S1–S20 (small subunit) bezeichnet werden. Die häufig stark basischen ribosomalen Proteine enthalten neben einer globulären Domäne meist eine fingerförmige Verlängerung. Sie sind auf der Oberfläche der Ribosomen angeordnet, wobei ihre Fingerdomänen in das Innere der Ribosomen reichen und dort mit der rRNA interagieren. Die Sequenz vieler ribosomaler Proteine ist während der Evolution (molekulare Evolution) nicht besonders gut erhalten geblieben. Überraschend viele scheinen für die Ribosomenfunktion nicht unbedingt erforderlich zu sein. Vermutlich haben diese Proteine eher die Aufgabe, die rRNA-Funktion zu erleichtern, indem sie die wichtigsten funktionellen Sequenzen der verschiedenen RNAs stabilisieren oder in Position halten. – Bestimmte Antibiotika, wie u.a. Puromycin, Chloramphenicol, Cycloheximid und Streptomycin, binden spezifisch an Ribosomen vom 70S- bzw. 80S-Typ und blockieren dadurch bestimmte Translationsschritte ( Ribosomen ). Die Antibiotika/Ribosom-Interaktionen sind recht spezifisch, an vielen ist nachweislich rRNA beteiligt. Eine genaue Analyse der molekularen Vorgänge der Wirkung der Antibiotika könnte zum Verständnis der zunehmenden Antibiotika-Resistenzen beitragen. Hemmstoffe von 80S-Ribosomen sind Ribosomen-inaktivierende Proteine, wie das Ricin. Biochemie (Geschichte der), differentielle Zentrifugation, Gentechnologie, Monosom, Prä-Proteine, Prä-Pro-Proteine, Protoribosomen, Ribosomenbindungsstelle, RNP-Welt, Wittmann (H.-G.); Ribosomen , Transkription – Translation, Zelle.
H.K./H.Z./S.Kl.
Lit.: Spirin, A.S.: Ribosome structure and protein synthesis. Benjamin 1986.
Ribosomen
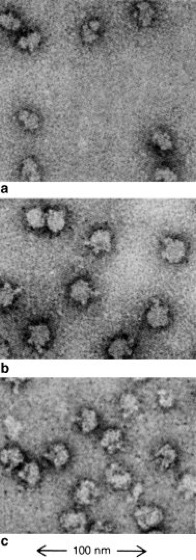
Abb. 1: Elektronenmikroskopische Aufnahmen von a kleinen, b großen ribosomalen Untereinheiten, c ganzen 70S-Ribosomen
Ribosomen
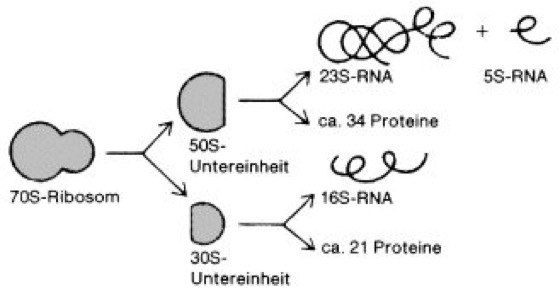
Abb. 2: Dissoziation von Ribosomen zu Untereinheiten und zu RNA- und Proteinkomponenten am Beispiel eines bakteriellen 70S-Ribosoms:
In Medien geringer Mg2+-Ionen-Konzentrationen zerfallen 70S-Ribosomen in die beiden unterschiedlich großen 50S- und 30S-Untereinheiten. Diese können im Reagenzglas weiter zu RNA- und Proteinketten zerlegt werden, die, einzeln isoliert, in ihren strukturellen Eigenschaften (Nucleotid- bzw. Aminosäurezusammensetzung, Primär- und Sekundärstrukturen, wechselseitige Bindungen usw.) charakterisierbar sind. Zur Größe und Komponentenanzahl anderer Ribosomen vgl. Tab .
Ribosomen
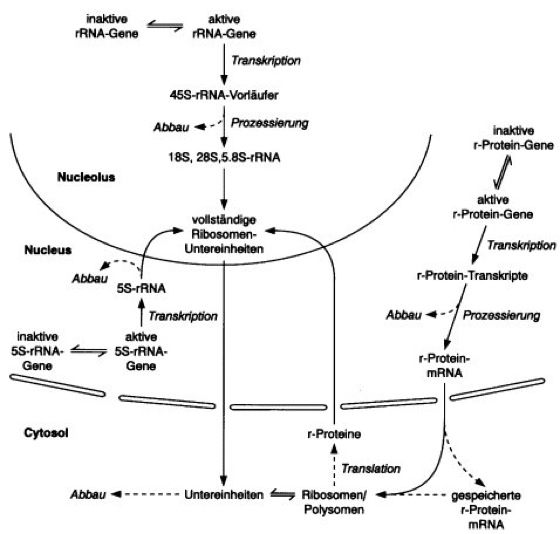
Abb. 3: Die Biogenese von funktionstüchtigen, aus vielen unterschiedlichen Komponenten zusammengesetzten Ribosomen setzt die Koordination einer Vielzahl von Synthesen und schließlich den Zusammenbau aller Bausteine voraus. Jeder der in der Abb. dargestellten Teilschritte kann als ein Ort möglicher Regulation für den Aufbau der Ribosomen angesehen werden und steht über Regelkreise mit den anderen Schritten in Verbindung. An der Biogenese der Ribosomen sind alle 3 eukaryotischen Genklassen und die zugehörigen RNA-Polymerasen beteiligt (im wesentlichen: die „großen“ rRNA-Gene und RNA-Polymerase I, Protein-codierende Gene und RNA-Polymerase II, die 5S-rRNA-codierenden Gene und RNA-Polymerase III). Außerdem erfordert der Zusammenbau der Ribosomen mehrfach einen Transport von Komponenten durch die Kernmembran (Kerntransport von Proteinen).
Ribosomen
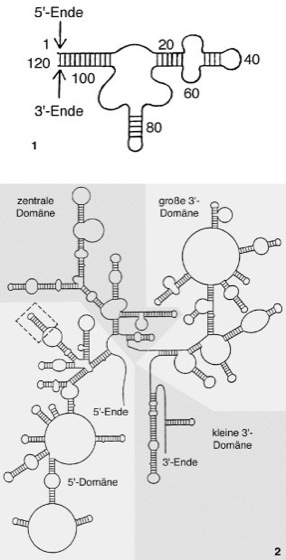
Abb. 4: Sekundärstrukturen von ribosomaler RNA.
1 Sekundärstruktur von 5S-rRNA. Die Ziffern geben die ungefähren Positionen der aus 120 Nucleotiden aufgebauten Kette an. 2 Sekundärstruktur von 16S-rRNA aus Escherichia coli. Die aus 1542 Nucleotiden aufgebaute Kette faltet sich durch intramolekulare Basenpaarungen in 4 kovalent verbundene Domänen (5'-Domäne: Position 1–560, zentrale Domäne: Position 560–920, große 3'-Domäne: Position 920–1400, kleine 3'-Domäne: Position 1400–1542). Die Basenpaarungen erfolgen sowohl zwischen näher benachbarten Sequenzen, die dann nur durch eine oder wenige ungepaarte „Schleifen“ getrennt sind, als auch zwischen zum Teil mehrere hundert Positionen entfernten Sequenzen, wie z.B. in der Nähe von Domänengrenzen. Da diese RNA besonders stark konserviert ist, gilt sie als Paradebeispiel der molekularen Evolution. Die Sekundärstrukturen von RNAs der kleinen ribosomalen Untereinheiten anderer Organismen zeigen eine ähnliche Grundstruktur, wobei aber im Einzelfall ganze Stamm-Schleifen-Elemente „amputiert“ bzw. eingefügt sind. So fehlt z.B. bei Chloroplasten-16S-rRNA aufgrund einer 23bp-Deletion im 16S-rRNA-Gen die durch Strichelung im oberen Teil der 5'-Domäne hervorgehobene „Haarnadel“-Struktur. Eine noch komplexere, aus 6 Domänen zusammengesetzte Sekundärstruktur wurde auch für 23S-rRNA postuliert.
Ribosomen
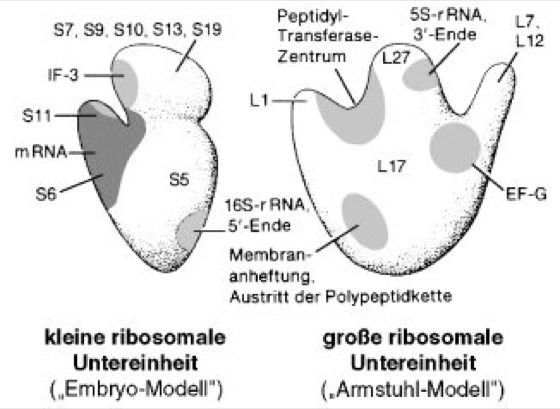
Abb. 5: Ribosomale Untereinheiten.
Die aus elektronenmikroskopischen Aufnahmen abgeleiteten, räumlichen Modelle der großen und kleinen ribosomalen Untereinheiten von Escherichia coli. Gezeigt ist auch die Lokalisation einzelner ribosomaler Proteine (S5, S6, S7, S9, S10, S11, S13 und S19 auf der kleinen Untereinheit und L1, L7, L12, L17 und L27 auf der großen Untereinheit), wie man sie durch die im Elektronenmikroskop erkennbare Bindung entsprechender Antikörper für fast alle ribosomalen Proteine auf bestimmte Bereiche annähernd eingrenzen kann. Ferner sind gezeigt die Lokalisation des 5'-Endes von 16S-rRNA und des 3'-Endes von 5S-rRNA, die Bindestellen für mRNA, Initiationsfaktor IF-3 und Elongationsfaktor EF-G, das Peptidyl-Transferase-Zentrum und die Austrittsstelle der Polypeptidketten. Bei der Bildung von 70S-Ribosomen lagert sich die kleine Untereinheit in (fast) Querlage (d.h. etwa 70° gegen den Uhrzeigersinn gedreht) im zentralen Bereich der großen Untereinheit an.
Ribosomen
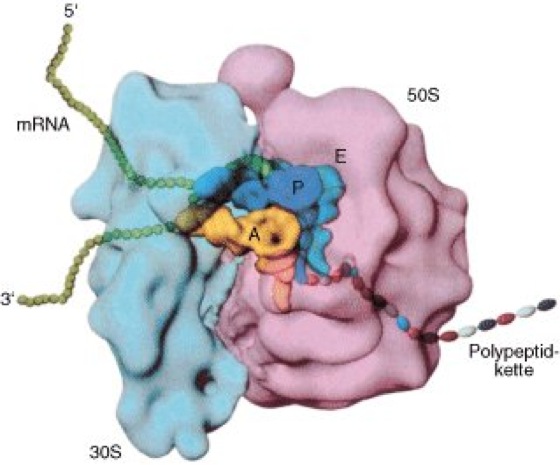
Abb. 6: Dreidimensionale Struktur des Ribosoms von Escherichia coli: Dei Modell zeigt den gesamten ribosomalen Komplex (70S), bestehend aus der kleinen (30S, hellblau) und großen (50S, rosa) Ribosomenuntereinheit; weiterhin sind die 3 Aminoacyl-tRNA-Moleküle (orange, blau, hellblau) dargestellt, die in den A-, P- und E-Positionen des Ribosoms vorliegen. Die mRNA (grüne Kette) gleitet nahe an den Anticodonschleifen der tRNA-Moleküle entlang, die nascierende Polypeptidkette liegt innerhalb eines Tunnels der großen Untereinheit, dessen Eingang sich etwa 10–15 Å vom 3'-aminoacylierten Ende der tRNA-Moleküle befindet. (Auflösung ca. 25 Å; Modell wurde nach kryoelektronenmikroskopischen Untersuchungen abgeleitet.)
Ribosomen
Struktur und Zusammensetzung von Ribosomen
|
| |||||
| Größe (Durchmesser in nm) | 20–24 | 30 | 21 | 15–20 | |
| Relative Teilchenmasse (in Mega-Dalton = 1,66·10–18 g) | 2,6–2,8 | 3,8–4,5 | 3,1 | 3 | |
| Sedimentationskonstanten: | |||||
| intaktes Ribosom | 70S | 80S | 70S | 80S (Ciliaten) 70–75S (Pilze) 70–80S (Pflanzen) 55–60S (Wirbeltiere) | |
| große und kleine Untereinheiten | 50S und 30S | 60S und 40S | 50S und 30S | – | |
| rRNAs aus: | |||||
| großer Untereinheit | (21–)23S 5S | (24–)28S 5,8S 5S | 23S 5S 4,5S (keine 4,5S bei Algen) | 24–26S, 16S bei Säugern 5S (keine 5S bei Pilzen und Tieren) | |
| kleiner Untereinheit | 16S | (16–)18S | 16S | 18,5S 12S bei Säugern | |
| Kettenlängen von rRNAs (Anzahl von Nucleotiden) | E.coli 2904 (23S) 120 (5S) 1541 (16S) | Rattenleber 4700 (28S) 160 (5,8S) 120 (5S) 1900 (18S) | Mais-Chloroplasten 2900 (23S) 121 (5S) 95 (4,5S) 1491 (16S) | menschliche Mitochondrien 1559 (16S) 954 (12S) | |
| Massenverhältnisse RNA/Protein | 3/2 | 1/1 | 1,13/1 | 0,43/1 (Tiere) | |
| Anzahl von r-Proteinen* große Untereinheit | 34 (E.coli) | ca. 40–50 | 26–34 | 36–40 | |
| kleine Untereinheit | 21 (E.coli) | ca. 30 | 20–25 | 30–33 |
* Die Proteine der großen (engl. large) Untereinheit werden nach der relativen Molekülmasse fortlaufend mit L1 (Protein mit der größten Molekülmasse), L2, L3 usw. bis L34 (Protein mit der kleinsten Molekülmasse bei Escherichia coli) beziffert. Analog ist die Bezifferung der Proteine der kleinen (engl. small) Untereinheit durch S1, S2, S3 usw. bis S21 (bei Escherichia coli)
Schreiben Sie uns!