Lexikon der Biologie: Zellkern
Zellkern, Kern, Nucleus, Nukleus, Karyon, genetisches Steuerzentrum und größtes Organell der Zelle der Eukaryoten (Eucyte). Zu den allgemeinen Funktionen des Zellkerns gehören die Speicherung der genetischen Information (Erbinformation) in linearen DNA-Doppelsträngen (Desoxyribonucleinsäuren), die Replikation dieser DNA sowie die Synthese der verschiedenen RNA-Spezies (Ribonucleinsäuren) im Zuge der Transkription und deren Prozessierung. Die Trennung des Erbmaterials vom Rest der Zelle dient dem räumlichen Zusammenfassen verwandter biochemischer Reaktionen in Kompartimente. Für die korrekte Gleichverteilung des hier gespeicherten Erbguts auf 2 genetisch identische Tochterkerne sorgt im Verlauf des Zellzyklus die Mitose (Farbtafel). Anders ist dies bei der Meiose (Farbtafel), in deren Verlauf eine weitgehende Durchmischung (Rekombination) des Erbguts erreicht wird. Durch die meiotischen Kernteilungen entstehen aus diploiden (Diploidie) Zygoten bzw. deren mitotisch entstandenen Tochterzellen wieder haploide (Haploidie) Zellen (Reduktionsteilung). Manche Zellen verlieren im Zuge irreversibler Differenzierungsprozesse (Differenzierung) ihren Zellkern (z.B. Säuger-Erythrocyten, Siebzellen und Siebzellenglieder bei Samenpflanzen). Sie sind dann zu keiner weiteren Entwicklung mehr fähig und gehen ebenso wie experimentell kernlos gemachte Zellen nach einer begrenzten Überlebenszeit zugrunde. – Wichtigste Strukturkomponenten des Zellkerns ( vgl. Infobox 1 , vgl. Abb. 1 ) sind Chromatin, Nucleolen, Kern-Grundsubstanz und Kernhülle. Als Chromatin bezeichnet man mit basischen Farbstoffen leicht anfärbbare (Färbemethoden) netzartige Strukturen des Interphase-Zellkerns, die sich zu Beginn von Mitose und Meiose zu den für Chromosomen charakteristischen Strukturen verdichten. Das sind, biochemisch gesehen, filamentöse Komplexe aus DNA und einer Vielzahl unterschiedlicher Proteine (Desoxyribonucleoprotein-Komplex, nucleärer DNP-Komplex). Prokaryoten besitzen keinen Zellkern (Protocyte), und ihre zirkuläre DNA (ringförmige DNA) ist nicht mit Histonen komplexiert. Dagegen ist das Eukaryoten-Chromatin grundsätzlich anders aufgebaut. Die DNA ist hier über die gesamte Länge mit Histonen komplexiert. Das in Form von Nucleofilamenten und Chromatinfibrillen elektronenmikroskopisch (Elektronenmikroskop) darstellbare Chromatin ist in Form von Nucleosomen organisiert. Verschiedene Histonproteine sind für diese „Perlenketten“-Struktur des Chromatins verantwortlich. Sie bewirken auch das Auftreten supranucleosomaler Überstrukturen und regulieren damit den Kondensationsgrad des Chromatins (Chromatinorganisation [Abb.]). Den Histonen und Nicht-Histonen (Nicht-Histon-Proteine, HMG-Proteine) werden regulatorische Funktionen bei der Transkription zugeschrieben. In neueren Untersuchungen konnte nachgewiesen werden, daß Myosin, ein Protein, das zusammen mit Actin für die Muskelbewegung (Muskelkontraktion) verantwortlich ist, auch an der Transkription beteiligt ist. Hier gleitet ein Strang, die DNA, an einem Enzym, der RNA-Polymerase, vorbei. Im Interphasekern (Arbeitskern) ist das Chromatin gegenüber den kompakten Metaphase-Chromosomen stark aufgelockert (Euchromatin). Es stellt den Bereich hoher Transkriptionsaktivität dar. Nur das Heterochromatin liegt auch im Interphasekern in Form dichter Chromozentren in kondensierter Form vor. Diese Unterscheidung in Eu- bzw. Heterochromatin ist schon seit langem bekannt. – Die meist sphärischen Nucleolen oder Kernkörperchen (Durchmesser 2–5 μm) sind die auffälligsten Einschlüsse des Zellkerns und aufgrund ihres kompakten Aufbaus und ihrer hohen Dichte auch im Licht-Mikroskop deutlich sichtbar. Hier findet die Synthese der Prä-Ribosomen statt (an denen jedoch noch keine Translation ablaufen kann). Die Nucleolen bilden sich am Ende der Mitose (in der Telophase), ausgehend von sekundären Einschnürungen bestimmter Chromosomen (sog. Satellitenchromosomen), die man als Nucleolus-Organisator-Regionen bezeichnet (Nucleolus). Da keine Proteinsynthese im Zellkern ablaufen kann, müssen viele Proteine nach ihrer Synthese im Cytoplasma in den Kernraum verlagert werden (Kerntransport von Proteinen). Diese sog. Nucleoproteine (z.B. Histone, DNA-RNA-Polymerasen) lassen sich ausschließlich hier nachweisen. – In den letzten Jahren wurden immer mehr regulatorische Proteine gefunden, die abhängig von Signalen (Steroidhormone, Licht, Entwicklungssignale) in den Kern eintransportiert werden und entweder selbst als Transkriptionsfaktoren wirken oder die Aktivität von Transkriptionsfaktoren steuern. In Abwesenheit des Signals werden diese Proteine durch zumeist noch unbekannte Retentionsfaktoren im Cytoplasma zurückgehalten. Daneben gibt es einen geregelten Export von solchen Proteinen aus dem Kern hinaus. Entscheidend für die Richtung des Proteintransports sind die mit dem kleinen G-Protein Ran (Ran-Proteine, Guanosintriphosphatasen) assoziierten Proteine (z.B. RanGAP), die an der Kernhülle (s.u.) sitzen und dort einen steilen Gradienten zwischen der GDP- (Guanosin-5'-diphosphat) und der GTP-haltigen (Guanosin-5'-triphosphat) Form von Ran aufrechterhalten. – Zellkerne sind meist kugelförmig. Bei Höheren Pflanzen ließ sich jedoch inzwischen über konfokale Laser-Scan-Mikroskopie zeigen, daß der Zellkern sehr oft einem Schweizer Käse ähnelt und zahlreiche Einbuchtungen aufweist, die in extremen Fällen sogar durch den gesamten Zellkern hindurchreichen können. Man vermutet, daß so eine größere Oberfläche und damit ein besserer Molekülfluß zwischen Kernplasma und Cytoplasma erzielt werden kann. – Die Grundsubstanz des Zellkerns ist eine amorph erscheinende Masse, die Chromatin und geformte Genprodukte enthält, und setzt sich aus einer großen Zahl unterschiedlich löslicher Proteine (Nuclear-Sol, früher Karyolymphe genannt; Kernplasma) sowie einem filamentösen Kernskelett aus Strukturproteinen (Nuclear-Gel, Nuclear-Matrix) zusammen. Als Hauptprotein des Nuclear-Sols enthalten tierische Zellkerne Nucleoplasmin, ein aus 5 gleichen Untereinheiten (relative Molekülmasse je 29.000) gebildeter Komplex. Nach Ergebnissen von Studien an Krallenfröschen (Xenopus) ist Nucleoplasmin u.a. maßgeblich an Aufbau und Modellierung des Chromatins während früher Entwicklungsphasen beteiligt. Nucleoplasmin ist weit verbreitet, in seiner Aminosäuresequenz konserviert (molekulare Evolution); es fehlt in inaktiven Zellkernen. – Das Kernskelett umfaßt etwa 1/10 aller nucleären Proteine (Kernproteine) und gliedert sich in Nuclear-Lamina, internes Kerngerüst und Nucleolarskelett. Die Nuclear-Lamina liegt der Innenfläche der Kernhülle an (Membranskelett) und setzt sich bei Wirbeltieren aus 3 untereinander ähnlichen hydrophoben Hauptproteinen zusammen (Lamine A, B und C). Die Lamine sind einerseits sehr eng mit der inneren Membran der Kernhülle verbunden, weisen andererseits jedoch auch starke Bindungstendenzen zum Chromatin auf. Darauf beruht offenbar die Strukturordnung des Chromatins im Interphasekern. Vor einer Mitose (Prophase) werden die Lamine stark phosphoryliert (Phosphorylierung), sie werden löslich, und die Kernhülle bricht zusammen. Hierbei vesikulieren die Membranen der Kernhülle und finden sich als ER-ähnliche Vesikel innerhalb des Cytoplasmas. Am Ende der Mitose (Telophase) sammeln sich die Lamine wieder unter Dephosphorylierung an den Chromosomenoberflächen sowie an der Innenseite der neu entstandenen Kernhülle. Bei Pflanzen, aber auch bei Hefe konnten trotz intensivster Bemühungen weder Gene für Lamine noch für ihre Bindepartner nachgewiesen werden. Die Struktur des pflanzlichen Zellkerns wird daher vermutlich über einen anderen Mechanismus aufrechterhalten. – Die Kernhülle (irreführende Bezeichnung „Kernmembran“) gewährleistet die strikte Trennung von aktivem Chromatin und allen anderen Zellkompartimenten (Kompartimentierung). Diese Perinuclearzisterne ist wohl die phylogenetisch älteste Ausbildungsform des eukaryotischen Endomembransystems ( vgl. Abb. 2 ). Sie unterscheidet sich vom übrigen endoplasmatischen Reticulum, in das sie meist an mehreren Stellen übergeht, durch ihren unmittelbaren Kontakt mit Chromatin und durch die Ausbildung von Kernporen (Durchmesser 50–70 nm). Die Kernporenkomplexe sind dynamische Strukturen, die in der Prophase verschwinden und bei der telophasischen Neubildung der Kernhülle wieder auftauchen. Der Makromolekültransport zwischen Cytoplasma und Kernplasma erfolgt ausschließlich durch die Porenkomplexe (RNA und RNP [Ribonucleoproteine] vom Zellkern ins Cytoplasma, alle nucleären Proteine in umgekehrter Richtung). Die Porenfrequenz (Porenkomplexe pro μm2) steigt mit Aktivität und Größe des Zellkerns ( vgl. Infobox 2 ) an (Kernporenkomplex [Tab.]). – Bedeutende Forschungen über den Zellkern leisteten unter den frühen Biologen u.a. R. Brown, R.J.W. Feulgen, O.W.A. Hertwig, G.A. Klebs, R.A. von Kölliker, J.F. Miescher, M.J. Schleiden. Cytologie, differentielle Zentrifugation, Evolution der Eucyte, fraktionierte Zentrifugation, Karyolyse, Kernfarbstoffe, Kernlokalisationssignal, Kern-Plasma-Relation, Kerntransplantation, mikroskopische Präparationstechniken, Pyknose, Zelltheorie; Photosynthese I , – Transkription – Translation , Zelle .
B.L./S.Kl.
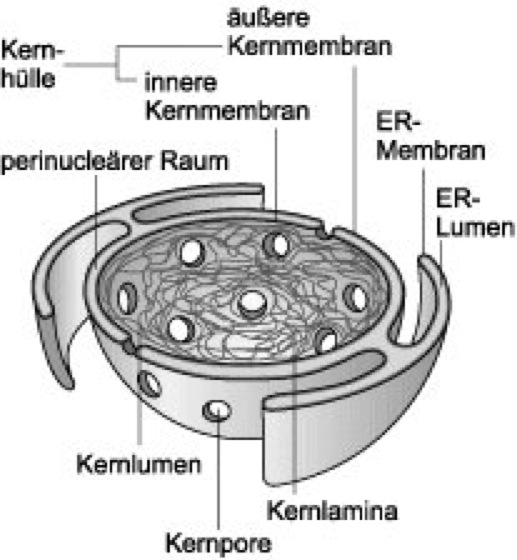
Zellkern
Abb.1: Räumliche Darstellung des Zellkerns.
Der Zellkern ist von einer Membranzisterne, der Kernhülle, umschlossen, an deren Innenseite sich die Kernlamina befindet, die als Teil des Kernskeletts dem Zellkern seine Form verleiht. Das plasmatische Innere des Zellkerns wird als Kernplasma oder Nucleoplasma bezeichnet. Hier befindet sich das Chromatin. Im Zellkern läuft nicht nur die Replikation der DNA ab, so daß bei der Mitose genetisch identische Tochterkerne entstehen, sondern hier erfolgt auch die Transkription der genetischen Information sowie die Prozessierung der dabei entstandenen RNA-Moleküle. Aus diesem Grund kann der Zellkern auch als die „Schaltzentrale“ der Zelle bezeichnet werden. – Die räumliche Trennung von Prozessen, die wie die Transkription ausschließlich im Zellkern ablaufen, und solchen, die wie die Translation nur im Cytoplasma lokalisiert sind, erfordert Transportvorgänge über die Kernhülle. Kleine Moleküle können dabei mittels Diffusion durch die Kernporen gelangen, wohingegen der Transport großer Moleküle energieabhängig ist. Da die Proteinsynthese nur im Cytoplasma erfolgen kann, müssen Kernproteine in den Zellkern importiert werden (Kerntransport von Proteinen). Mittels einer sog. NLS-Sequenz (NLS von engl. nuclear localization signal; Kernlokalisationssignal) werden Proteine, die für den Zellkern bestimmt sind, von cytoplasmatischen Faktoren (Importine) erkannt, um anschließend als Komplex an Kernporen anzudocken. In analoger Weise sorgt eine NES-Sequenz (NES von engl. nuclear export signal) dafür, daß Proteine den Zellkern verlassen. Diejenigen Proteine, die wie einige, an Primärtranskripte bindende Proteine im Zellkern verbleiben, zeichnen sich hingegen durch eine NRS-Sequenz (NRS von engl. nuclear retention signal) aus.
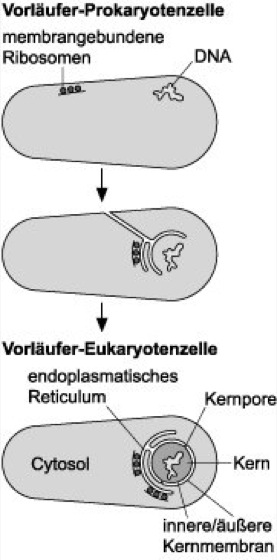
Zellkern
Abb. 2: Hypothese zur Entstehung des Zellkerns durch eine die DNA umgebende Membraneinstülpung: Während der Evolution von der Protocyte zur Eucyte kann der Zellkern dadurch entstanden sein, daß die Plasmamembran die an dieser anhaftende DNA durch eine Einstülpung allmählich umschlossen hat. Durch eine vollständige Abschnürung von der Plasmamembran kann ebenfalls das endoplasmatische Reticulum entstanden sein.
Schreiben Sie uns!