Lexikon der Biochemie: Rekombination
Rekombination, genetische Rekombination, die Gesamtheit der Prozesse, die während der Meiose oder Mitose zur Bildung neuer Genkombinationen führt. R. ereignen sich häufig und in allen Organismen. Man unterscheidet zwei Formen der R.: die allgemeine R. und die spezialisierte bzw. ortsspezifische R. Zu letzterer gehören solche Rekombinationsformen, die keine homologen Regionen in den Eltern-DNA-Molekülen benötigen, wie z.B. 1) Chromosomenumlagerungen (innere R.), 2) Umlagerungen von Genen für Vertebratenimmunglobulin und T-Zellrezeptoren, sowie 3) Transposition (Transposon) und 4) Erzeugung eines lysogenen Zustands durch Inkorporation von Bakteriophagen-DNA in das Wirtschromosom (Phagenentwicklung). Der vorliegende Artikel befasst sich ausschließlich mit der allgemeinen R., die als ein Austausch zwischen einem beliebigen Paar homologer Sequenzen in parentalen DNA-Molekülen definiert werden kann.
Bei der meiotischen (sexuellen) R. können sowohl Gene rekombiniert werden, die auf unterschiedlichen Chromosomen liegen (nicht gekoppelt), als auch Gene, die auf dem gleichen Chromosom lokalisiert sind (gekoppelt, Genkopplung). Nicht gekoppelte Gene können frei rekombiniert werden. Dies ist zurückzuführen auf die zufällige Orientierung der Bivalente und die zufällige Verteilung der Chromosomen während der Meiose sowie der zufälligen Gametenvereinigung während der Befruchtung. Der Vorgang gehorcht den mendelschen Regeln.
Gekoppelte Gene verhalten sich während der Meiose wie eine Einheit, d.h. sie können nicht frei rekombiniert werden.
Die somatische (parasexuelle) R. läuft während der Mitose ab, jedoch nicht während der Meiose. Bei höheren Organismen kommt die somatische R. selten vor, bei bestimmten Pilzen, bei Bakterien und Viren stellt sie jedoch den normalen Rekombinationsvorgang dar. Bakterien erreichen den heterogenetischen Zustand, der eine Vorbedingung für die genetische R. ist, durch Konjugation, Transduktion oder Transformation.
R. wird durch ein Crossing-over der beteiligten DNA-Stränge herbeigeführt. Die Intermediate, die hierbei auftreten, lassen sich anhand eines Modells erklären. Zwei ausgerichtete homologe DNA-Doppelstränge, die einen Bruch enthalten, überkreuzen sich und paaren sich mit ihrem komplementären Gegenstück. Anschließend werden die Brüche geschlossen. Alle Modelle für die gebildete viersträngige, überkreuzte Struktur der Heteroduplex (Holliday-Struktur) zeigen, dass alle Basen ohne sterische Spannung gepaart vorliegen. In der Abb. werden zwei mögliche Mechanismen zur Auflösung der Holliday-Struktur, d.h. die Spaltung der DNA-Ketten und die anschließende Bildung der beiden DNA-Doppelstränge, aufgezeigt.
In E. coli spielt das recA-Protein (352 Aminosäurereste; Produkt des recA-Gens) bei der R. eine Hauptrolle. RecA bindet spezifisch und kooperativ an einzelsträngige DNA, wobei ein filamentöser Komplex gebildet wird. Zur Einleitung des Austauschs homologer Segmente assoziiert dieses Nucleofilament dann mit doppelsträngiger DNA. Die Duplex-DNA wird anschließend rasch nach einer Sequenz abgetastet, die komplementär zur einzelsträngigen DNA ist. Während des Abtastvorgangs wird die Duplex durch einen ATP-abhängigen Prozess, der durch recA katalysiert wird, teilweise entwunden. Wenn eine komplementäre Sequenz angetroffen wird, paart sich diese mit der recA-bedeckten, einzelsträngigen DNA. Es findet ein Strangaustausch statt und eine gerichtete Strangwanderung (engl. branch migration) führt zur Bildung eines neuen Heteroduplexmoleküls sowie zu einem verschobenen Strang der einzelsträngigen DNA. Der verschobene Strang bildet eine D-förmige Schleife (D loop), der aus dem Hauptkörper der DNA herausragt.
Die Einzelstrangbrüche, die für die recA-Bindung notwendig sind, werden mit Hilfe des ATP-abhängigen Enzyms recBCD (Mr 330 kDa, Produkt der SOS-Gene recB, recC und recD) eingeführt. RecBCD windet und entwindet Duplex-DNA, wobei die Entwindung schneller verläuft als die Windung, so dass zwei einzelsträngige Schleifen gebildet werden. Diese beiden Schleifen werden größer, während das Enzym an der Duplex entlangwandert, bis es auf die Sequenz 3'GCTGGTGG5' trifft. Diese Chi-Sequenz kommt im E.-coli-Chromosom ungefähr einmal bei 10.000 Basen vor. RecBCD spaltet anschließend die Einzelstrangschleife 4-6 Nucleotide vom 3'-Ende der Chi-Sequenz entfernt. An der R. in E. coli sind darüber hinaus eine Vielzahl weiterer Proteine beteiligt.
Der Rekombinationsmechanismus in Eukaryonten wurde noch nicht so intensiv untersucht wie der in E. coli. Das Hefe-RAD51-Protein, das 30%ige Homologie zu recA aufweist, katalysiert ebenfalls ATP-abhängige DNA-Reparatur und R. Sein Komplex mit DNA ist unter dem Elektronenmikroskop praktisch identisch mit demjenigen von recA. RAD1-Homologe werden in einer Vielzahl von Tieren, einschließlich Hühnern, Mäusen und Menschen, gefunden. Daraus wird geschlossen, dass der Grundmechanismus der R. bei allen Organismen der gleiche ist. [R. Holliday Genet. Res. 5 (1964) 282-304; S.C. West Annu. Rev. Biochem. 61 (1992) 603-640; A.F. Taylor Cell 69 (1992) 1.063-1.065; R.M. Story et al. Nature 355 (1992) 318-325 (s. a. S. 367); E.H. Engelman Curr. Opin. Struct. Biol. 3 (1993) 189-197; M. Takahashi u. B. Norden Adv. Biophys. 30 (1994) 1-35; S.C. West Cell 76 (1994) 9-15; J. Lee u. M.J ayaram J. Biol. Chem. 270 (1995) 4.042-4.052; S.E. Corrette-Bennett u. S.T. Lovett J. Biol. Chem. 270 (1995) 6.881-6.885; M.M. Cox J. Biol. Chem. 270 (1995) 26.021-26.024; F. Maraboeuf et al. J. Biol. Chem. 270 (1995) 30.927-30.932]
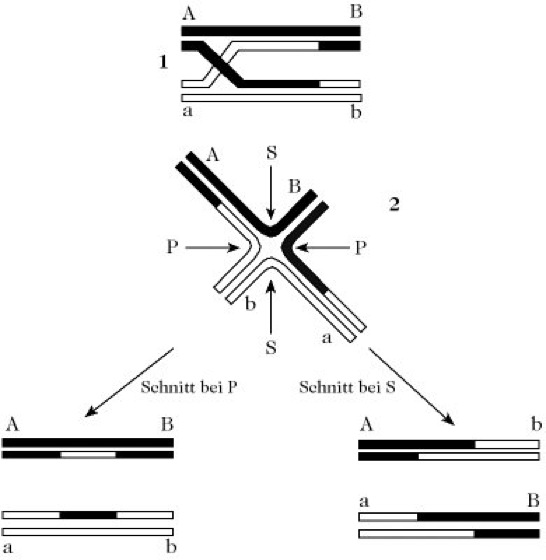
Rekombination. Auflösung des Crossing-over bei homologer Rekombination. 1) Die Holliday-Struktur: Es hat ein Crossing-over zwischen den DNA-Strängen stattgefunden und die Überkreuzungsstelle hat sich durch "branch migration" weiterbewegt. 2) Alternative graphische Darstellung der Vierwege-DNA-Kreuzung der Holliday-Struktur. Je nachdem, ob die Struktur bei P oder bei S gespalten wird, entstehen zwei unterschiedliche Paare rekombinanter Moleküle.
Schreiben Sie uns!