Kompaktlexikon der Biologie: Transkription
Transkription, der 1. Schritt der Genexpression, bei dem es, von bestimmten RNA-Viren abgesehen, zu einer DNA-abhängigen Synthese der Ribonucleinsäuren kommt, die durch RNA-Polymerasen katalysiert wird und zur Bildung von messenger-RNA, transfer-RNA, ribosomaler RNA und einer Reihe weiterer RNA-Spezies führt. Die T. ähnelt der DNA-Replikation (Replikation) dahingehend, dass eine matrizenabhängige Synthese von Nucleinsäuren aus Nucleosidtriphosphaten erfolgt; sie unterscheidet sich von dieser aber auch in wesentlichen Merkmalen: Bei der T. wird nur ein DNA-Strang (Matrizenstrang, codogener Strang, Template) und von diesem nur ein kleiner Teil in RNA umgeschrieben; hinzu kommt, dass RNA-Polymerasen die Fähigkeit zum Proof-reading fehlt. (DNA-Polymerasen)
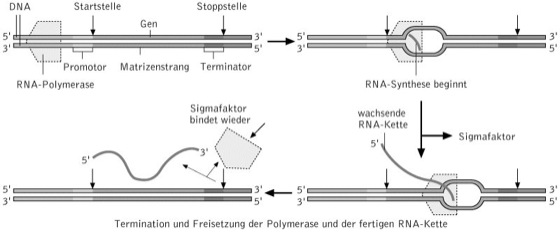
Zwischen der T. prokaryotischer und eukaryotischer Zellen bestehen, was die biochemischen Grundlagen der RNA-Synthese anbelangt, generell keine Unterschiede. Jedoch sind an der T. bei beiden Zelltypen verschiedene Strukturen und Proteinfaktoren auf unterschiedliche Weise wirksam. So kommt z.B. bei Eukaryoten mehr als eine RNA-Polymerase vor, sodass die verschiedenen RNA-Spezies durch unterschiedliche Enzyme synthetisiert werden. Hinzu kommt, dass die Verpackung eukaryotischer DNA in Chromosomen die T. beeinflusst. Allg. lässt sich die T. in drei Teilschritte unterteilen, die Initiation („Kettenstart“), bei der es zur Wechselwirkung des zu transkribierenden Gens mit der RNA-Polymerase kommt, die Elongation („Kettenverlängerung“) als Phase der RNA-Synthese und die Termination („Kettenabbruch“), die zur Beendigung der T.-Prozesse und Freigabe der RNA führt ( vgl. Abb. ).
Initiation. Zu Beginn der T. kommt es zu einer Erkennung bestimmter Sequenzmotive im Promotor eines Gens durch die RNA-Polymerase. Sie wird durch gerichtete Diffusion der RNA-Polymerase entlang der DNA-Doppelhelix möglich, wobei elektrostatische Wechselwirkungen sowie Interaktionen des Enzyms mit Wasserstoffdonoren und -akzeptoren der DNA auftreten. Bei Prokaryoten erfolgt die spezifische Erkennung mittels des so genannten Sigma-Faktors, bei eukaryotischen Zellen wird diese durch Transkriptionsfaktoren wie z.B. TBP (= TATA-Box bindendes Protein) vermittelt. Der Sigma-Faktor ist Bestandteil des bakteriellen RNA-Polymerase-Holoenzyms und erkennt spezifische Motive in der doppelsträngigen DNA. Bei diesen handelt es sich um Sequenzen, die als Minus-10-Region (Pribnow-Box) und Minus-35-Box bezeichnet werden, da sie 10 bzw. 35 Nucleotide stromaufwärts vom Transkriptionsstart, der das erste Nucleotid der RNA darstellt, lokalisiert sind. Im Anschluss an diese Prozesse deckt die RNA-Polymerase einen Bereich von –50 bis +20 Basenpaaren ab. Dieser so genannte geschlossene Komplex wird abschließend durch partielles Entwinden der DNA in den offenen Komplex überführt. Nach erfolgter Initiation dissoziiert der Sigma-Faktor von der RNA-Polymerase, die eine Konformationsänderung durchläuft und mit der RNA-Synthese beginnt.
Elongation. In dieser Phase bewegt sich die RNA-Polymerase in 5'-3'-Richtung an der DNA entlang, wobei es am Matrizenstrang (codogenen Strang) zur RNA-Synthese in 5'-3'-Richtung kommt. Innerhalb des DNA-RNA-Proteinkomplexes, der Transkriptionsblase, kommt es vor dem 3'-Ende der entstehenden RNA zu einer Entwindung von jeweils 17 Basenpaaren der DNA, der eine Verdrillung folgt, nachdem die RNA-Polymerase einen DNA-Abschnitt abgelesen hat. Durch die wiederentstehende Doppelhelix wird die RNA aus der zunächst entstandenen DNA-RNA-Helix verdrängt.
Termination. Die T. erfolgt so lange, bis die RNA-Polymerase auf eine Terminatorsequenz stößt, an der es zum Anhalten der RNA-Synthese kommt und sich die RNA und das Enzym vom Komplex lösen. Bei Prokaryoten sind zwei verschiedene Arten der Termination bekannt, die sich dahingehend unterscheiden, ob an ihr der Rho-Faktor beteiligt ist. Bei der Rho-unabhängigen Termination verursacht ein GC-reicher Bereich die Entstehung einer Haarnadel-Struktur (engl. hairpin loop) in der RNA, wobei angenommen wird, dass sich die T.-Rate aufgrund einer langsameren Entwindung der GC-reichen Sequenz ebenfalls verlangsamt und die Bildung der Haarnadel-Struktur fördert ( vgl. Abb. ). An den GC-reichen Bereich schließt sich stets eine aus sechs Nucleotiden bestehende polyA-Sequenz an. Die auf Ebene der RNA vorhandene polyU-Sequenz bildet mit der polyA-Sequenz der DNA nur relativ schwache Wechselwirkungen, sodass es schließlich zu einem Lösen der RNA vom Matrizenstrang und somit zur Beendigung der T. kommt. Bei der Rho-abhängigen Termination bindet der Rho-Faktor zunächst an die entstehende RNA und bewegt sich durch ATP-Hydrolyse auf der RNA entlang zur Polymerase, wo es zur Entwindung der DNA-RNA-Helix und letztlich zur Auflösung der Transkriptionsblase kommt. Bei Eukaryoten erfolgt die Termination der T. analog zur faktorenunabhängigen Termination. Im Unterschied zu detaillierten Kenntnissen über die Struktur eukaryotischer Promotoren und Trankriptionsfaktoren, liegen über eukaryotische Terminatoren weniger Informationen vor.
Die differenzielle Genexpression, d.h. die Tatsache, dass Zellen Gene zu unterschiedlichen Entwicklungsstadien und Zeiten exprimieren, wird bei Eukaryoten durch Transkriptionsfaktoren, Enhancer und Silencer kontrolliert, wohingegen bei Prokaryoten verschiedene Sigma-Faktoren existieren, die eine spezifische Anbindung an bestimmte Promotoren ermöglichen. Während der nach seinem Molekulargewicht von 70 kDa genannte σ70-Faktor für die T. eines breiten Spektrums von Genen verantwortlich ist, kontrollieren andere Sigma-Faktoren die Expression bestimmter Gene bzw. Gruppen von Genen. Besonders gut untersucht ist z.B. der σ32-Faktor, der spezifisch für die Promotoren von Hitzeschockgenen ist. In gleicher Weise erkennt σ54 Gene, die im Zusammenhang mit der Nutzung alternativer Stickstoffquellen stehen und σ32 Gene, die für die Bildung von bestimmten Proteinen der Flagellenstruktur zuständig, die an chemotaktischen Prozessen beteiligt sind (Chemotaxis).
An die T. schließen sich je nach Art der entstandenen RNA unterschiedliche Prozesse an (Prozessierung, Spleißen, Translation): ( vgl. Abb. )
Transkription: Schema der Transkription bei Prokaryoten

Transkription: Haarnadelförmige Sekundärstruktur im 3'-nichtcodierenden Bereich einer prokaryotischen mRNA. Links ist ein Stoppcodon für die Translation (UAA) zu erkennen
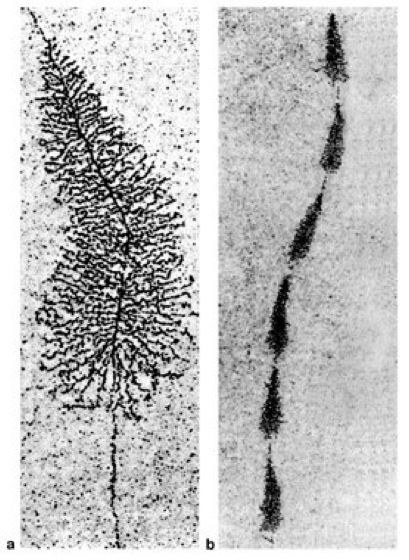
Transkription: Elektronenmikroskopische Aufnahmen, die die Transkription von ribosomaler RNA (rRNA) entlang eines rDNA-Gens zeigen. a Mit zunehmender Entfernung vom Transkriptionsstart (oben) sind die rRNA-Transkripte gut zu erkennen, da die Transkription durch viele RNA-Polymerasen gleichzeitig erfolgt. b Bei geringerer Vergrößerung ist die Anordung der für rRNA codierenden Gene gut zu erkennen
Schreiben Sie uns!